新闻动态NEWS
您现在所在的位置: 首页» 新闻动态 News魏文胜老师荣获第二十三届 “吴阶平-保罗·杨森医学药学奖”
2023.06.092023年6月8日,第二十三届“吴阶平-保罗·杨森医学药学奖”(简称吴杨奖)获奖者名单公布。经过严格的初评和终评,吴杨奖最终评选出基础医学、临床医学、药学、公共卫生领域15位优秀医药卫生工作者为获奖人。课题组魏文胜老师因其在基因编辑技术、高通量功能性基因组学及新型核酸治疗技术等领域取得的突出成绩而榜上有名。
魏文胜课题组致力于开发新型基因编辑工具,发展高通量功能基因组学技术和创建新型核酸治疗平台,并据此发展新的疾病治疗策略。
作为基因编辑研究领域的国际领军人物,魏文胜教授带领其研究团队在国际上率先建立并发展了一系列全基因组范围的高通量功能性筛选策略,发明了LEAPER等具有我国自主知识产权的新型基因编辑底层技术,极大促进了基因编辑及相关应用领域的发展。同时,魏文胜教授团队首次报道了环状RNA疫苗技术并据此开发了针对新冠病毒的环状RNA疫苗,为生物医学研究和疾病治疗提供了新的手段。
资料链接:
为促进我国医药卫生事业的发展,激励广大医药卫生工作者发扬严谨治学、求实创新精神,国家卫生健康委国际交流与合作中心利用社会资源与强生公司所属的西安杨森制药有限公司于1994年共同设立了吴阶平-保罗•杨森医学药学奖(简称吴杨奖),表彰、奖励在医药卫生领域努力钻研并独立作出突出贡献、被社会及同行广泛认可的60岁及以下优秀医药卫生工作者。
1994年至今,吴杨奖以其科学、严格的评选程序,严肃、认真的评审态度,确立了在医药卫生领域的声誉和地位,成为我国医药卫生工作者努力争取的一项殊荣。
供稿人:牛煦然
Nature Biotechnology | 本课题组报道新型线粒体碱基编辑器
2023.05.22线粒体作为细胞的“能量中心”在细胞生命活动过程中扮演着重要角色,它还是细胞核外存储遗传信息的另一细胞器。据MITOMAP数据库统计,在已确认的97种线粒体遗传疾病中,93种由点突变引起,因此使用碱基编辑工具修正这些突变具有重要意义。然而线粒体中的电子传递将质子从线粒体基质排出,使得基质带有强负电荷,这阻碍了具有相同负电荷核酸(如CRISPR系统的sgRNA)的进入1。全蛋白基础的基因编辑工具ZFN和TALEN可以在定位信号的引导下进入线粒体,之前有研究报道在小鼠线粒体中可以据此靶向敲低突变的基因组2,3。然而,敲低线粒体基因组不能治疗纯合的线粒体突变,也不能主动改变线粒体基因组的碱基构成。
基于CRISPR-Cas系统开发的单碱基编辑在治疗基因组点突变引起的遗传疾病方面显示出巨大潜力。碱基编辑器中使用的脱氨酶都是单链DNA脱氨酶,在Cas9和sgRNA的作用下,目标DNA双链中的非靶向链暴露出来,为单链DNA脱氨酶实现有效碱基转换提供了必要条件4。与CRISPR-Cas系统相比,锌指蛋白(ZF)和转录激活因子样效应子(TALE)只具有结合DNA双链的活性,却无法解开DNA双链5。因此,简单将单链DNA脱氨酶与ZF或TALE结合无法实现对DNA的有效碱基编辑6。
2020年,Joseph Mougous实验室和David Liu实验室利用一种能够作用于双链DNA的脱氨酶DddA开发出了线粒体单碱基编辑器,首次实现了线粒体基因C->T的碱基编辑7。2022年Jin-Soo Kim实验室使用DddA和TadA8e组合,进一步实现了线粒体基因组A->G的碱基编辑8。然而,左二伟实验室和伊成器实验室随后发现DddA系统存在比较严重的脱靶效应9,10,特别是DddA与CTCF存在相互作用,会产生细胞核基因组的非特异性编辑10。
2023年5月22日,北京大学魏文胜课题组在Nature Biotechnology杂志在线发表题为 “Strand-selective base editing of human mitochondrial DNA using mitoBEs” 的研究论文,报道了一种名为mitoBEs的全新线粒体单碱基编辑工具,该工具不依赖于DddA系统。mitoBEs不仅能够高效地实现A->G或C->T的单碱基编辑,还具备选择性地编辑特定链的能力,这是DddA系统所不具备的。此外,研究人员未观察到明显的脱靶现象。
由于目前发现的除DddA之外的DNA脱氨酶,都只能作用于单链DNA,整个研究基于这样的假设:在靶向位点产生瞬时的单链DNA,可以为所有“普通” 脱氨酶提供有效反应底物。因此,在TALE系统提供靶向的基础上,研究者整合了切口酶(nickase)和脱氨酶(deaminase),成功建立了高效的TALE版本的单碱基编辑器,实现了线粒体基因组的碱基编辑(mitochondrial DNA base editors, mitoBEs)。使用含有线粒体定位信号的TALE-MutH和TALE-TadA8e(V106W)靶向线粒体基因组,可以实现有效的A->G编辑(图1)。对切口酶MutH的突变研究,可以大幅度突破其序列偏好,将可编辑范围提高10倍以上。此外,无识别序列偏好的切口酶Nt.BspD6I(C)和FokI-FokI(D450A),也能够有效应用于mitoBEs系统(图2)。
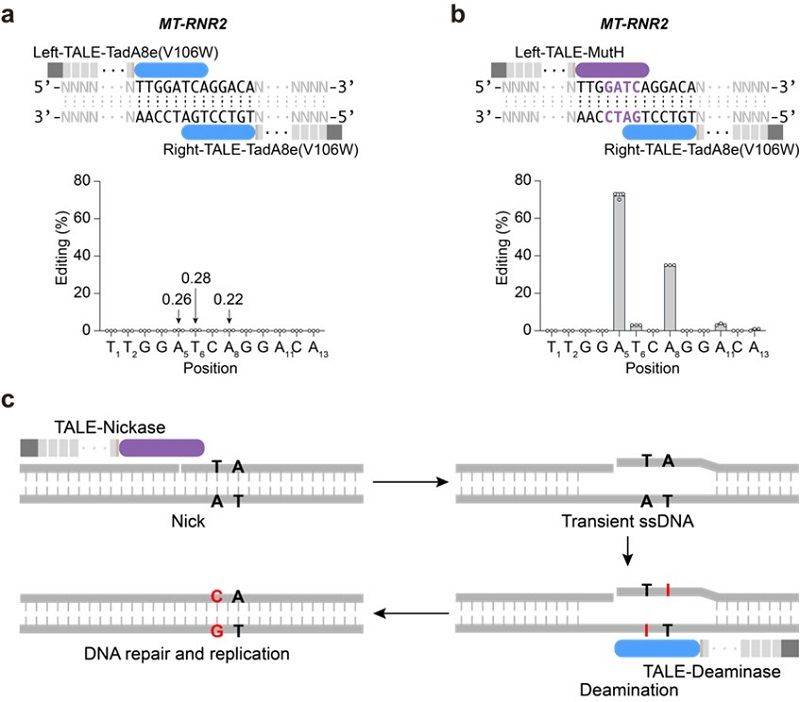
图1: 引入nickase来实现链选择性DNA编辑及mitoBE工作模型
图2: 筛选可以用于mitoBE系统的无识别序列限制的nickase
除了A->G方向的编辑,TALE-MutH和TALE-rAPOBEC1-2×UGI的结合可以实现C->T的高效碱基转换。与DdCBE相比,mitoBEs具有链选择性偏好。此外,通过全基因组测序,mitoBEs在线粒体和细胞核中都没有检测到严重的脱靶编辑,证明其具有高度特异性和安全性。
研究者进一步尝试这一新型编辑器在疾病治疗中的应用。Leber遗传性视神经病变 (Leber's hereditary optic neuropathy, LHON) 是一种由线粒体基因突变引起的急性眼部疾病,患者均为成年人。研究者利用环状RNA编码的mitoBEs实现了高效线粒体DNA链选择性碱基编辑,成功建立了疾病模型。随后,针对LHON患者来源的细胞,使用环状RNA编码的mitoBEs在目标位点实现了约20%的编辑效率,修复后的细胞具有更高的ATP含量和氧化呼吸水平,表明mitoBEs对线粒体遗传疾病具有治疗潜力。这是首次通过碱基编辑方式修正了线粒体的致病突变(图3)。由于理论上这一新技术能够修正大多数线粒体疾病突变(图3),这就为治疗这些危害重大的疾病提供了有希望的治疗方法。此外,该技术方案也适用于细胞核基因组的碱基编辑,对相关疾病治疗提供了潜力巨大的新工具。

图3: 使用mitoBE纠正LHON病人来源细胞的线粒体基因突变
北京大学魏文胜课题组博士后伊宗裔、博士研究生张小雪为论文的共同第一作者,唐玮、于莹、魏晓旭和张雪对文章也作出了重要贡献。该研究项目得到了国家重点研发计划、国家自然科学基金重点项目、北京市科委生物医学前沿创新推进项目、北大-清华生命科学联合中心、昌平实验室和中国博士后科学基金的支持。
文章链接: https://doi.org/10.1038/s41587-023-01791-y
1 Gammage, P. A., Moraes, C. T. & Minczuk, M. Mitochondrial Genome Engineering: The Revolution May Not Be CRISPR-Ized. Trends Genet 34, 101-110, doi:10.1016/j.tig.2017.11.001 (2018).
2 Bacman, S. R. et al. MitoTALEN reduces mutant mtDNA load and restores tRNA(Ala) levels in a mouse model of heteroplasmic mtDNA mutation. Nat Med 24, 1696-1700, doi:10.1038/s41591-018-0166-8 (2018).
3 Gammage, P. A. et al. Genome editing in mitochondria corrects a pathogenic mtDNA mutation in vivo. Nat Med 24, 1691-1695, doi:10.1038/s41591-018-0165-9 (2018).
4 Anzalone, A. V., Koblan, L. W. & Liu, D. R. Genome editing with CRISPR-Cas nucleases, base editors, transposases and prime editors. Nat Biotechnol 38, 824-844, doi:10.1038/s41587-020-0561-9 (2020).
5 Deng, D. et al. Structural basis for sequence-specific recognition of DNA by TAL effectors. Science 335, 720-723, doi:10.1126/science.1215670 (2012).
6 Yang, L. et al. Engineering and optimising deaminase fusions for genome editing. Nat Commun 7, 13330, doi:10.1038/ncomms13330 (2016).
7 Mok, B. Y. et al. A bacterial cytidine deaminase toxin enables CRISPR-free mitochondrial base editing. Nature 583, 631-637, doi:10.1038/s41586-020-2477-4 (2020).
8 Cho, S. I. et al. Targeted A-to-G base editing in human mitochondrial DNA with programmable deaminases. Cell 185, 1764-1776 e1712, doi:10.1016/j.cell.2022.03.039 (2022).
9 Wei, Y. et al. Mitochondrial base editor DdCBE causes substantial DNA off-target editing in nuclear genome of embryos. Cell Discov 8, 27, doi:10.1038/s41421-022-00391-5 (2022).
10 Lei, Z. et al. Mitochondrial base editor induces substantial nuclear off-target mutations. Nature 606, 804-811, doi:10.1038/s41586-022-04836-5 (2022).
兔飞猛进,大展宏兔| Wei Lab举行2023新年午餐会
2023.01.12
今天是农历腊月二十一,还有十天就将迎来2023农历癸卯兔年。恰值京城初雪时分,纷飞的瑞雪携声声祝福洒落燕园,又给今日添上了几分喜色。
在这个辞旧迎新的节点上,实验室全体成员一同举行了一场热闹的新年午餐会。伴随着主持人的开场,一阵又一阵的欢声笑语渐渐调动起了大家的热情劲儿;魏老师对过去的一年做了总结,对新年的到来进行了展望,并对实验室的全体成员表示了感谢;周老师和武老师也依次为大家送上了新年的祝福。大家一起共享了丰盛的午餐,笑声、启瓶声、倒酒声声声入耳,整个实验室洋溢着着新年的快乐气息。
2022已经过去,农历虎年也渐行渐远。历添新岁月,春满旧山河。兔年的钟声即将敲响,愿实验室的全体成员能在新的一年里兔飞猛进,大展宏兔!

供稿人:牛煦然
恭喜实验室多位同学获得2020-2021年研究生奖励奖学金
2022.10.10根据《北京大学学生奖励评选办法》(校发〔2018〕234号)、《北京大学学生奖励评选办法实施细则》(学工发〔2018〕25号)、《北京大学奖学金评审办法》相关规定,经过个人申请、班级民主素质测评、研究生导师推荐、研究生专业初评、学院审核等程序,本实验室多名同学获得了2021-2022学年度研究生奖励和奖学金,具体名单如下:
三好学习标兵:唐慧贤
三好学生:潘倩,徐艺源,席蕾
优秀科研奖:马华峥,田峰,王春慧,拉毛切忠,陈奉
兴业奖学金:拉毛切忠,田峰,王春慧,徐艺源
北京大学三等奖学金:潘倩
国家奖学金:唐慧贤
向各位同学表示热烈的祝贺!
Wei Lab在第二十一届LAB杯背后的故事
2022.06.29来自前方记者(队长潘倩)不正经报道。
一年一度的LAB杯体育联赛终于在毕业季的“感时花溅泪”中落下帷幕。Wei Lab的勇士们发挥出色,一举拿下总冠军,奖金高达688万(划掉)元。接下来,就让我们一起走进比赛前前后后里那些欢欢笑笑的故事吧!
PART 0 确定队名
大赛在即,确定一个响亮的队名,也是迈出了成功的一大步。关于队名,大家展开了激烈的头脑风暴。最终牛煦然同学的“魏稳胜队”从众多候选名称中脱颖而出。从此,“魏稳胜”队诞生了!

热烈的取名讨论
PART 1 两人三足
在一个风和日丽的初夏午后,两人三足比赛在五四小操场上拉开了序幕。在不断变换队形和勇于尝试后,“魏稳胜”战队终于以“13.95 s”的成绩夺得亚军。虽然和冠军失之交臂略有遗憾,但这已是我们的历史最好战果!

呈现人字形排开的战队
PART 2 乒乓球
由小黄帽徐艺源同学带领的乒乓球队一路披荆斩棘,最终荣获第四名。杨四金同学“伊藤式”发球让大家啧啧称奇。小黄帽多个漂亮的接发和高质量扣球得分让我们见识到了专业选手的风采。

小黄帽大战小姑凉

球来了

抓狂的瞬间

好兄弟

兵乒球比赛现场
PART 3 台球
台球队由台球小王子巍少、蓝胖子哆啦峰梦、靓仔和奉哥组成。四个人的完美配合让我们成为赢得冠军的殿军。

传说中战胜冠军的一杆球

摆烂瞬间

思考人生的奉哥
PART 4 羽毛球
羽毛球咱们也不赖,拉毛队长RP爆炸,带领“WenYu复兴队”,一路过五关斩(躲)六将,最终挺进四强,获得第四名。

“WenYu复兴”战队
PART 5 排球:
和赵进东实验室组成的“不好意思在打球”战队在排球比赛中荣获亚军。(虽然只有两支队伍!但,我们是亚军!)

排球比赛合照
PART 6 篮球
常年腿部受伤的张巍同学和满身腱子肉的冯子琦同学率队出征。最终,毫无悬念地摘得桂冠。

帅气的篮球少年们和C位的巍少
PART 7 庆功
最终,Wei Lab取得了总成绩第一名的好成绩!向参赛的各位同学表示最热烈的祝贺!而实验室也以“炸鸡盛宴”为本届LAB杯画上了圆满的句号。
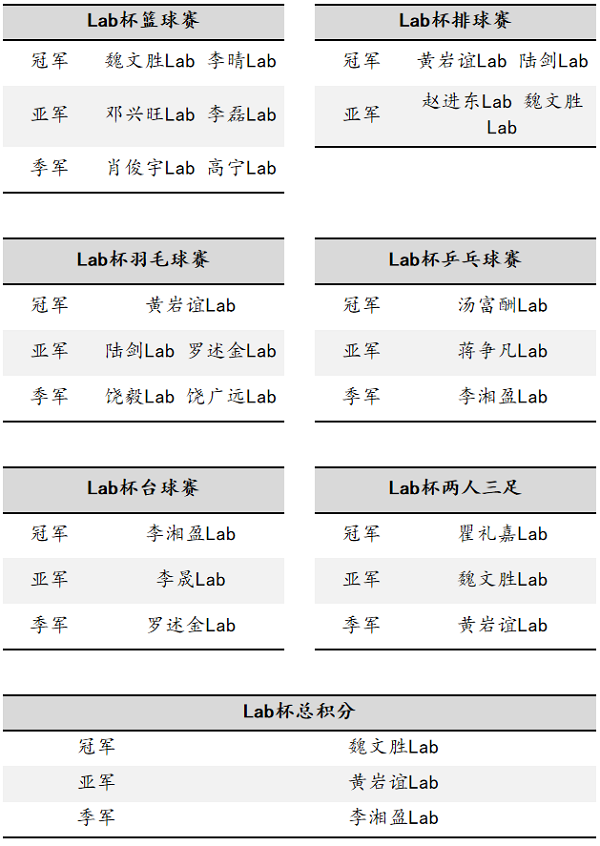
LAB杯体育联赛结果总览

炸鸡饕餮盛宴
PART 8 番外篇1.0
由于疫情缘故,50米赛跑和拔河比赛取消了,有不少小伙伴未能参与其中。咱留点遗憾,明年再战。
PART 9 番外篇2.0
以下为认真训练中的各位:

训练照1

训练照2

训练照3
供稿人:潘倩
编辑:牛煦然
祝贺张心怡同学和张芷瑄同学分别获得张景钺奖和沈同奖
2022.06.212022年6月,经自主申请和北京大学生命科学学院组织答辩,Wei Lab 2016级博士生张心怡获得“2022年生命科学学院优秀毕业研究生张景钺奖”,2018级本科生张芷瑄获得“2022年生命科学学院优秀毕业生沈同奖”。
“优秀毕业研究生张景钺奖”和“优秀毕业生沈同奖”两项荣誉以北大生科历史上两位老先生命名,是生命科学学院授予研究生和本科生的最高荣誉。祝贺两位同学在今年夏天分别为自己的研究生和本科生涯都画上了圆满的句号!
附:

张心怡,北京大学生命科学学院2016级博士生,在校期间多次获得校长奖学金、国家奖学金、优秀科研奖等奖励和北京大学优秀毕业生、北京大学三好学生等荣誉。在本实验室学习期间,她建立了蛋白质关键氨基酸定位技术PASTMUS并将该技术应用于蛋白质定向进化;同时在新冠疫情爆发后,也积极参与到新冠病毒的研究工作中,发现了新冠病毒激活宿主天然免疫的新机制。此外,她还曾担任北京大学学生风雷街舞社副社长一职,也积极参与学生志愿工作。张心怡同学毕业后将在产业界开启新的人生起点,愿张心怡同学永葆燕园情怀,在生物医药领域发挥北大人的光彩!

张芷瑄,北京大学生命科学学院2018级本科生,同时获得国家发展研究院经济学双学位。在校期间获得过北京市优秀毕业生、北京大学优秀毕业生、北京大学三好学生、北京大学红楼艺术奖等多项荣誉;同时积极参加文体活动,曾获2019年世界合唱大奖赛国际金奖等奖项。2020年加入北京大学魏文胜实验室,目前已通过4+4本博直通项目考核,将于今年秋天在Wei Lab继续踏上人生的新旅程,愿张芷瑄同学在实验室继续取得更新更好的成绩!
供稿人:牛煦然
祝贺张心怡、何苑、岳頔和伊宗裔同学获得博士学位
2022.06.202022年5月,实验室张心怡、何苑、岳頔和伊宗裔四位同学顺利通过学位论文答辩。恭喜几位同学获得博士学位!

同时,祝贺张心怡同学获得“北京大学优秀毕业生”的荣誉以及“北京大学生命科学学院优秀毕业研究生张景钺奖”!祝贺伊宗裔同学获评“北京大学优秀毕业生”和“北京市普通高等学校优秀毕业生”!

又是一个丰收的夏季,而夏天又开启了太多新的故事。时间的流逝是那么地令人猝不及防,但美妙的缘分让我们在燕园和Wei Lab相聚,在人生中属于青春年华的这段美好时光里一起乘坐着追逐梦想的巴士,看日出日落,看月圆月缺,看繁星璀璨,看那些我们记忆中最美丽的风景。
愿从Wei Lab走出的毕业生同学们此去繁花似锦,再相逢依旧如故!胸中永远澎湃着的未名水声,伴你们走过生命中一个又一个轮回的四季;心里永远荡漾着的燕园情怀,许你们踏遍天南海北也依旧青春少年!
供稿人:牛煦然
祝贺潘倩同学和徐艺源同学获得2022-2023年度校长奖学金
2022.06.20根据《北京大学博士研究生校长奖学金管理办法》,经过生命科学学院组织的答辩和评审工作,本实验室2018级博士研究生潘倩同学和2019级博士研究生徐艺源同学获得了2022-2023年度校长奖学金。向徐艺源同学表示热烈祝贺!
潘倩同学和徐艺源同学在课程学习、学术研究等方面均有优异表现,相信在今后的学习和科研中,将更好的发挥示范带动作用,鼓励本实验室的同学们更加专注学业精进,实现品学兼优、全面发展。
Nucleic Acids Research | 本课题组与合作者报道非编码调控元件 能够稳定染色质三维结构及细胞稳态
2022.04.11人类基因组的非编码序列中,启动子和增强子是调控基因表达的重要功能元件。大多数情况下,激活的增强子会与近端启动子发生直接互作。同时,也存在一部分增强子能够通过染色质成环、蛋白质寡聚化及RNA聚合酶II结合增强子沿染色质追踪等方式与线性基因组中的远端启动子互作来发挥调控功能,增强子与启动子的远距离互作体现了调控元件活性能够影响三维染色质结构。近年来,越来越多的研究表明增强子与启动子互作对于形成特定的三维空间结构至关重要,如转录因子和聚合酶在细胞核中不是均一分布,而倾向于聚集在转录活跃或染色质致密的区域;转录过程如转录延伸也能够影响染色质结构。这些在空间聚集成簇且转录活跃的调控元件是否能够维持大范围染色质结构,目前还没有系统性的研究。
2022年4月7日,本课题组与加州大学圣地亚哥分校(UCSD)王巍课题组在Nucleic Acids Research上联合发表了题为 “Regulatory elements can be essential for maintaining broad chromatin organization and cell viability” 的研究论文,该研究基于前期工作中建立的EpiTensor算法(Zhu et al. Nature Communications 2016)发现了基因组中活跃的增强子/启动子互作中心位点,将该类位点称为hotspot。通过CRISPR/Cas9系统对非编码hotspot增强子位点进行靶向片段删除进而进行高通量功能性筛选,发现了一系列位点与细胞生长和存活密切相关。该研究首次发现该类增强子位点的删除能够改变大范围染色质结构,通过同时影响多个非必需基因表达所产生的协同效应来影响细胞稳态。
由于目前能够有效发现增强子/启动子互作的高分辨率Hi-C数据较少,本研究利用分辨率可达200-bp的EpiTensor算法,预测了73个正常细胞系和5个癌细胞系/组织中的活跃增强子/启动子的三维互作,获得了增强子与启动子、增强子与增强子以及启动子与启动子的强烈互作位点hotspot。我们发现该调控元件互作网络(Regulatory element interaction network,REIN)为小世界网络,即对节点的随机破坏不受影响但对于高度值节点的靶向破坏十分敏感(图1A-B)。为了探究hotspot位点是否参与稳定染色质三维结构,该研究利用实验室建立的CRISPR/Cas9系统介导的pgRNA片段删除策略,在K562细胞中对选取的751个非编码hotspot增强子位点进行高通量功能性筛选,发现了43个影响细胞存活或增殖的重要功能区(称为必需hotspot)(图1C)。其中,hotspot_10_25(chr10: 74,123,469–74,1248,68)位点的删除能够导致K562细胞产生显著的细胞死亡或生长抑制,而不影响其他多种癌细胞(图1D)。
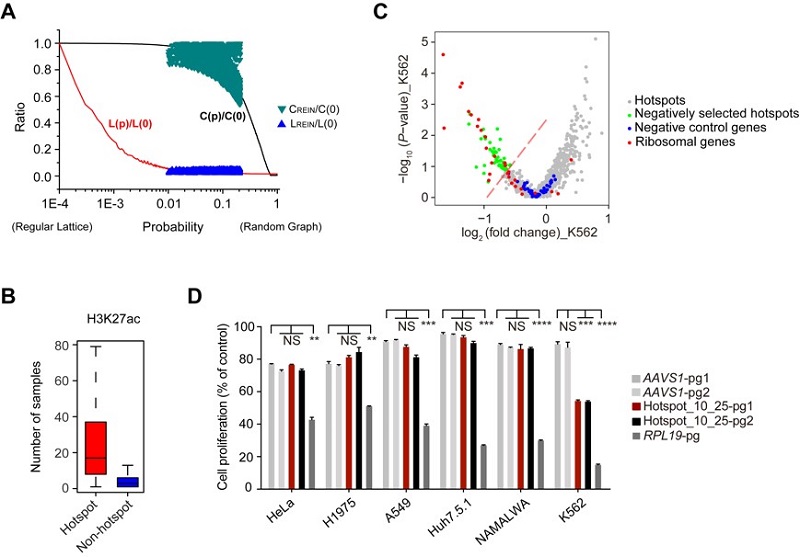
图1 hotspot位点的发现及高通量功能性筛选
为了探究hotspot位点删除是否影响染色质结构,该研究选取hotspot_10_25位点进行进一步研究,该位点不与任何必需基因存在互作。Hi-C分析发现该位点删除后其附近6–8 Mb区域的有效直径(effective diameter)和模块化打分(modularity score)发生显著改变,并观察到一些与该hotspot邻近区域发生互作的区域(chr10: 11–17 Mb)也发生改变(图2A-C)。与之相一致地,在chr10: 12–14 Mb区域也发现了拓扑结构相关结构域(TAD)发生破坏。这些染色质结构的改变导致多个基因的增强子与启动子互作发生改变,包括CELF2, RSU1, FAM149B1和CCAR1等(图2D),这些被影响的增强子和启动子并不局限于被删除的hotspot位点附近,甚至可以远达62 Mb。由此表明,hotspot位点的删除可以影响大范围染色质结构,而不限于其线性基因组的邻近区域。单细胞测序进一步发现,该hotspot位点的删除会激活一系列细胞凋亡相关基因的表达,并且可以同时影响TAD内与之存在互作的多个非必需基因的表达,研究证明多基因表达改变的协同效应能够显著影响细胞存活(图2E)。该研究首次揭示了非编码调控元件增强子在稳定染色质三维结构中的重要性,而不局限于已知的通过增强子与启动子的直接互作来进行调控。
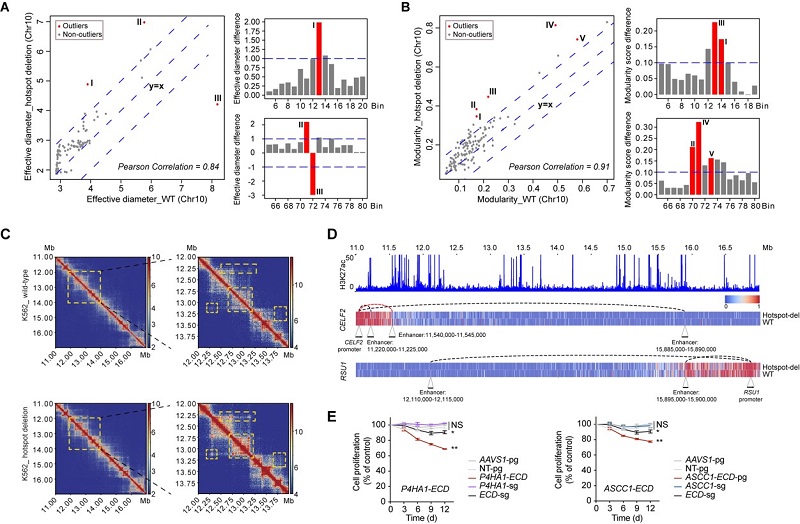
图2 hotspot位点删除导致大范围三维染色质结构和多基因表达的同时改变
值得一提的是,2021年11月UCSD王巍和本课题组合作在Science Advances上首次报道了无任何表观遗传信号的非编码位点hub能够稳定染色质三维结构并维持细胞存活(Ding et al., Science Advances 2021)。通过对hub和hotspot这两类非编码位点进行系统性挖掘、高通量筛选及深入功能解析,将进一步丰富对基因组非编码区域的功能阐述,并逐步揭示其在维持三维基因组结构中的重要作用。
北京大学刘莹博士、加州大学圣地亚哥分校丁博博士、博士生郑丽娜和北京大学许萍博士为该论文的共同第一作者,北京大学魏文胜教授和加州大学圣地亚哥分校王巍教授为该论文的共同通讯作者。该研究项目得到了国家自然科学基金、北京市科委生命科学前沿创新培育项目、北京未来基因诊断高精尖创新中心、北大-清华生命科学联合中心、中国博士后科学基金、加州再生医学研究所(CIRM)以及美国国立卫生研究院(NIH)的支持。
原文链接:https://doi.org/10.1093/nar/gkac197
Cell | 本课题组报道环状RNA技术平台以及据此开发的新型抗新冠病毒疫苗
2022.04.022022年3月31日,本课题组在Cell杂志上在线发表题为 “Circular RNA Vaccines against SARS-CoV-2 and Emerging Variants” 的研究论文,首次报道了环状RNA疫苗技术平台,以及据此开发的针对新冠病毒及其一系列变异株的环状RNA疫苗。该项研究中制备的针对新冠病毒德尔塔变异株的环状RNA疫苗(circRNARBD-Delta)对多种新冠病毒变异株具有广谱保护力(图1)。
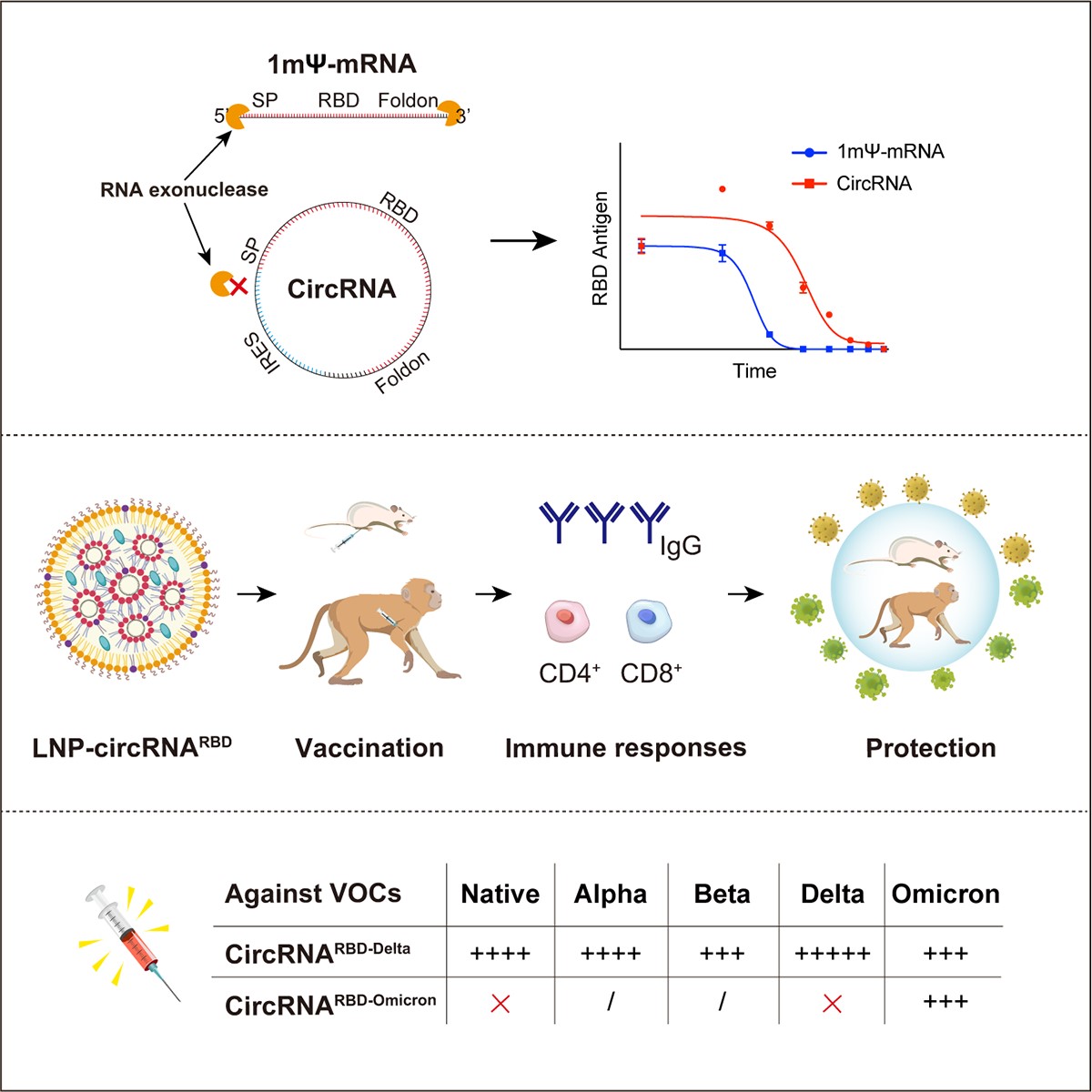
图1 新冠肺炎病毒circRNA疫苗研发示意图
作为近几年兴起的突破性医学技术,mRNA疫苗的基本原理是通过脂纳米颗粒(LNP)将mRNA导入体内来表达抗原蛋白,以刺激机体产生特异性免疫反应。2019年底新冠肺炎(COVID-19)爆发后,针对性的mRNA疫苗(Moderna mRNA-1273; Pfizer/BioNTech BNT162b2)在多种疫苗类型中脱颖而出。mRNA疫苗的修饰及递送技术均产生于国外机构,制约了我国mRNA疫苗及其治疗技术的发展和应用,因此亟需发展新型、高效的疫苗技术。与线性的mRNA不同,环状RNA分子呈共价闭合环状结构,不含5’-Cap和3’-polyA结构;且不需要引入修饰碱基, 其稳定性高于线性RNA。但是RNA的环化方法、纯化策略尚不成熟,其潜在的免疫原性对疫苗研发的影响并不清楚,诸多未知因素制约着环状RNA的研发应用。
魏文胜团队首先建立了体外高效制备高纯度环状RNA的技术平台,针对新型冠状病毒及其变异株,设计了编码新冠病毒刺突蛋白(Spike)受体结构域(RBD)的环状RNA疫苗。实验证明,该疫苗可以在小鼠和恒河猴体内诱导产生高水平的新冠病毒中和抗体以及特异性T细胞免疫反应,并可以有效降低新冠病毒感染的恒河猴肺部的病毒载量,显著缓解新冠病毒感染引起的肺炎症状(图2)
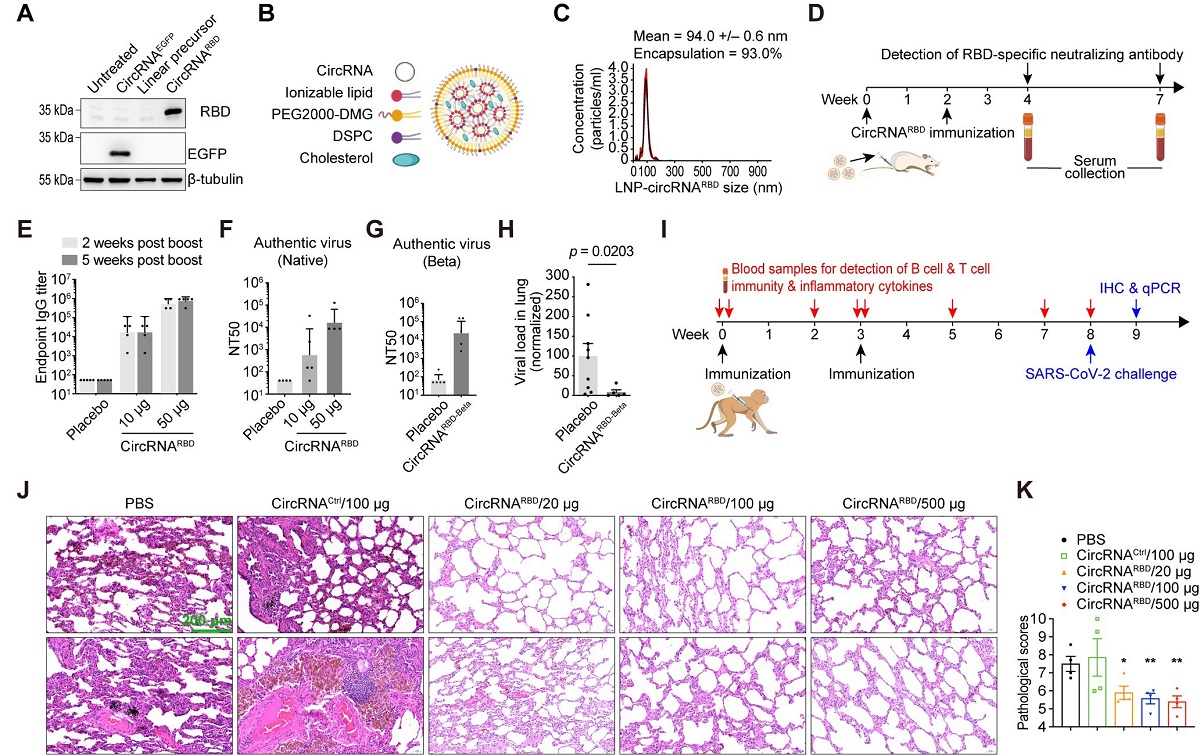
图2 CircRNA疫苗接种在小鼠和恒河猴体内提供了显著性保护
一系列的对比评估表明,与mRNA疫苗相比,circRNA疫苗具有以下特点或优势(图3):1)circRNA具有更高的稳定性,可以在体内产生更高水平、更加持久的抗原;2)circRNA疫苗诱导机体产生的中和抗体比例更高,可以更有效地对抗病毒变异,降低疫苗潜在的抗体依赖增强症(ADE)副作用;3)circRNA疫苗诱导产生的IgG2/IgG1的比例更高,表明其主要诱导产生Th1型保护性T细胞免疫反应,可以有效降低潜在的疫苗相关性呼吸道疾病(VAERD,Vaccine-associated enhanced respiratory diseases)副作用。
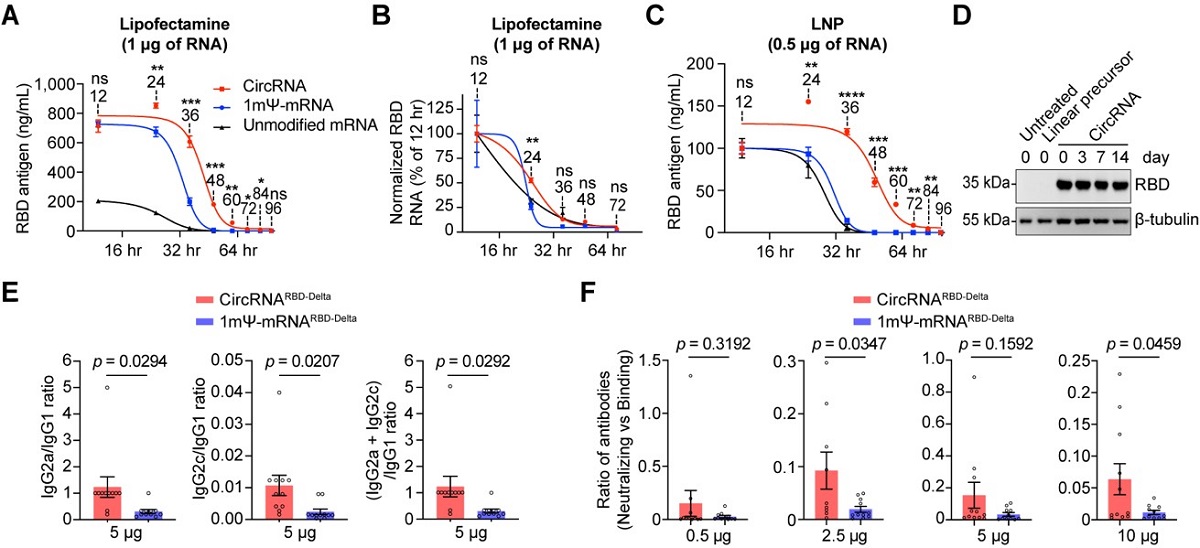
图3 CircRNA疫苗的特点和优势(相比于mRNA疫苗)
此外,在新冠病毒奥密克戎突变株被世界卫生组织列为值得关注的变异株(Variants of Concern,VOC)后,研究团队紧急启动了针对该突变株的环状RNA疫苗研发。在获得病毒序列信息的30天内,完成了从疫苗生产、小鼠免疫到有效性评估的全流程。研究发现,基于奥密克戎变异株的环状RNA疫苗(circRNARBD-Omicron)的保护范围狭窄,其诱导产生的抗体只能够中和奥密克戎变异株。而针对德尔塔变异株设计的环状RNA疫苗(circRNARBD-Delta)则可以在小鼠体内诱导产生广谱的中和抗体,有效中和包括奥密克戎株在内的多种新冠变异株(图4)。
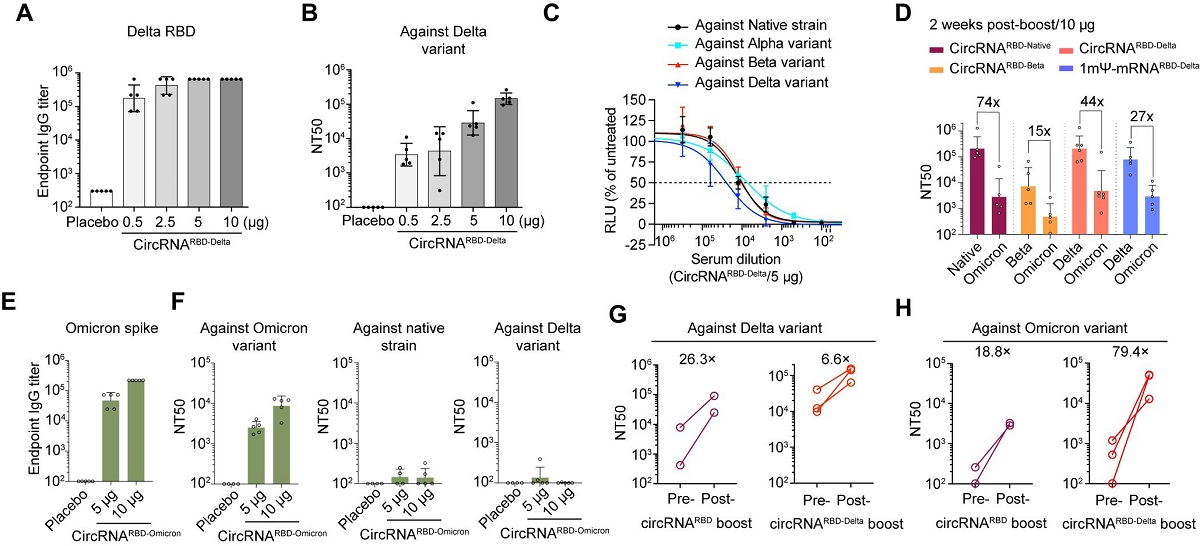
图4 针对新冠病毒德尔塔变异株设计的circRNARBD-Delta疫苗是一种具有广谱保护力的候选疫苗
以上结果表明,针对新冠病毒德尔塔变异株设计的circRNARBD-Delta疫苗是具有广谱保护力的新冠肺炎候选疫苗,该研究也为针对当前新冠变异株迅速传播的疫苗研发和接种策略提供了参考依据。同时,该项平台型技术的建立在感染性疾病、自身免疫病、罕见病以及癌症的预防或治疗中具有广泛的应用前景。
北京大学魏文胜课题组博士后璩良、博士研究生伊宗裔和沈勇为论文共同第一作者。本项研究获得了众多合作实验室的鼎力支持和帮助,包括北京大学谢晓亮教授/曹云龙研究员课题组,中国医学科学院/北京协和医学院王健伟教授课题组,中国医学科学院医学生物学研究所彭小忠教授课题组,中国食品药品检定研究院王佑春课题组及黄维金课题组。该研究项目得到了国家重点研发计划、国家自然科学基金重点及面上项目、北京市科委生物医学前沿创新推进项目、北京未来基因诊断高精尖创新中心、北大-清华生命科学联合中心以及传染病防治国家科技重大专项的基金支持。璩良博士获2020年度国家“博新计划”基金支持。
文章链接: https://www.cell.com/cell/fulltext/S0092-8674(22)00394-4
Nature Biotechnology|本课题组报道升级版RNA编辑技术:LEAPER 2.0
2022.02.11RNA编辑是近年来兴起的基因编辑技术。2019年,北京大学魏文胜课题组在Nature Biotechnology杂志报导了新型RNA编辑技术LEAPER1。与以CRISPR为基础的DNA或者RNA编辑技术不同,LEAPER仅需要在细胞中表达特殊设计的RNA(ADAR-recruiting RNA, arRNA)即可招募细胞中内源脱氨酶ADAR,实现靶向目标RNA中腺苷A→肌苷I(鸟苷G)的编辑。由于无需引入外源编辑酶或效应蛋白,避免了由此引起的递送以及相关的免疫原性等问题。另外作为RNA精准编辑工具,LEAPER不会引起基因组序列改变,在安全性方面具有优势。尽管LEAPER在科研和疾病治疗中具有可观的潜力,该技术还存在一定的局限:一是LEAPER利用的是内源编辑酶,其编辑效率会因此受限;另外,具有一定长度的arRNA可能使目标编辑位点邻近的碱基发生脱靶编辑,因此该技术亟需优化升级。
2022年2月10日,北京大学魏文胜课题组在Nature Biotechnology杂志发表“Engineered circular ADAR-recruiting RNAs increase the efficiency and fidelity of RNA editing in vitro and in vivo”的研究长文。该研究发现,通过优化表达载体中的启动子增强arRNA表达可以显著提升LEAPER系统的编辑效率,表明arRNA在细胞中的丰度对于编辑效率十分重要。然而线性arRNA在细胞内容易被降解的特点成为了制约因素。为了克服这一问题,课题组通过设计并运用可招募ADAR的环形RNA(circular ARAR-recruiting RNA, circ-arRNA),实现了编辑效率提升。
环形RNA没有5’或3’末端,可以避免核酸外切酶的切割,在细胞内相比于线性RNA具有更好的稳定性和更长的半衰期。研究发现,circ-arRNA能够维持较长时间的高水平表达。在多个内源转录本位点中,circ-arRNA平均编辑效率相比于线性版本提升了超过3倍,同时也可维持长达近半个月的有效编辑。通过腺相关病毒(AAV)递送,遗传编码的circ-arRNA可以在人的原代细胞和类器官中实现长时程的RNA编辑。另外,体外合成的circ-arRNA也可实现高效的靶向编辑,并且与遗传编码的circ-arRNA具有类似的特征。
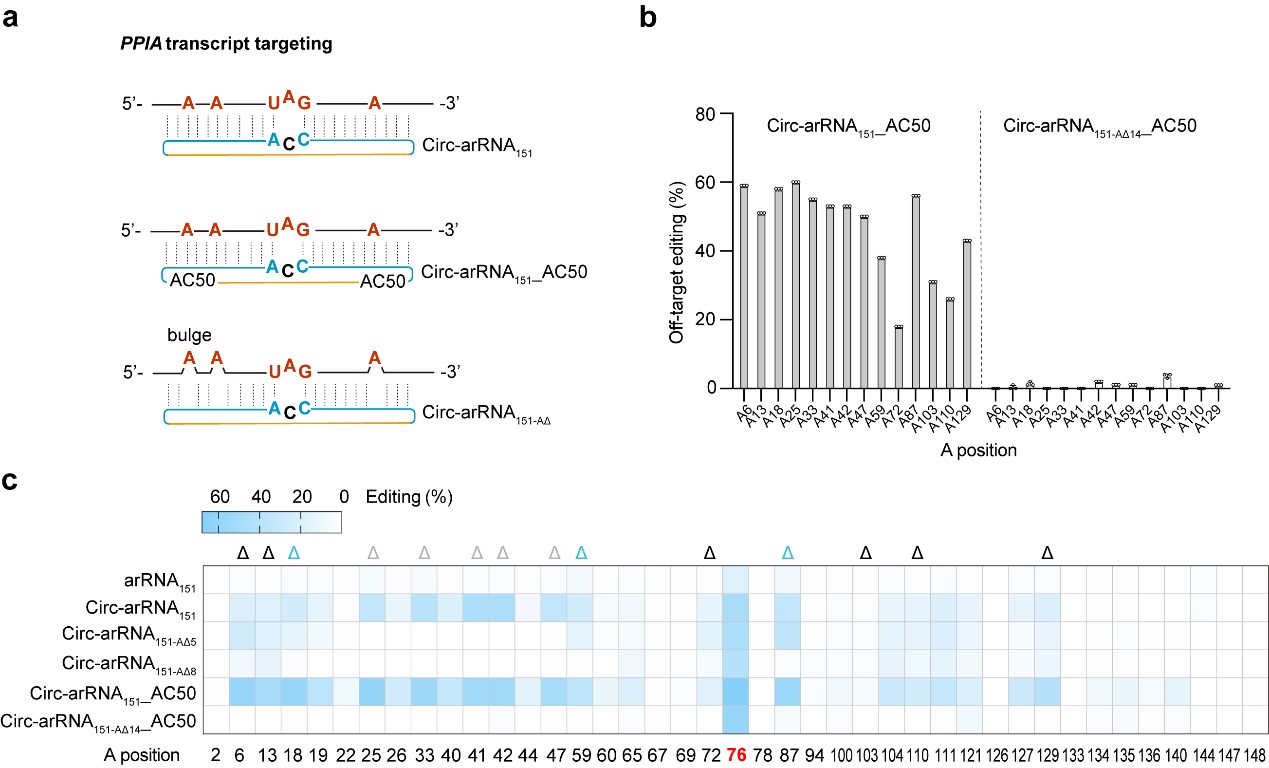
图一、Circ-arRNA在内源转录本上实现高效、精准的编辑
由于ADAR蛋白的底物为双链RNA,靶向RNA与circ-arRNA形成的双链区域内的腺苷酸会有不同概率的脱氨风险。消除这种邻近碱基的脱靶编辑(bystander off-target editing)是实现更加精准的单碱基编辑的关键。进一步研究发现,当删除arRNA或circ-arRNA上非靶向腺苷酸对面的核苷酸后,可以有效避免非靶向腺苷酸的编辑。据此重新设计的circ-arRNA在内源转录本上基本消除了双链RNA区域内目标转录本上的脱靶,同时维持了较高的精准靶向编辑效率。近期,Thorsten Stafforst课题组报道了名为CLUSTER的RNA编辑技术2,该技术虽然降低了邻近碱基的脱靶编辑效率,但是其靶向编辑效率仍处于和线性arRNA类似的水平。与此相比,circ-arRNA不仅提升了RNA编辑效率,还消除了邻近碱基的脱靶编辑,这一升级版技术被命名为LEAPER 2.0。

图二、LEAPER 2.0 — 通过工程化的circ-arRNA实现高效、精准的RNA编辑
接下来对LEAPER 2.0应用潜能的评估显示,circ-arRNA可以成功激活Wnt信号通路,修复TP53基因中的致病突变使其表达的p53恢复转录调节功能。使用AAV将circ-arRNA递送至Hurler综合征疾病模型小鼠体内,可以成功修复Idua致病突变并恢复IDUA的酶活。这些结果表明,LEAPER 2.0在科研和疾病治疗中具有令人期待的优势与潜能。
北京大学魏文胜课题组博士研究生伊宗裔(CLS)、博士后璩良和博士研究生唐慧贤(BIOPIC)为该论文的共同第一作者。该研究项目得到了国家重点研发计划、国家自然科学基金重点项目、北京市科委生物医学前沿创新推进项目、北京未来基因诊断高精尖创新中心、北大-清华生命科学联合中心以及中国博士后科学基金的支持。
文章链接:https://www.nature.com/articles/s41587-021-01180-3
1. Qu, L. et al. Programmable RNA editing by recruiting endogenous ADAR using engineered RNAs. Nat Biotechnol 37, 1059-1069 (2019).
2. Reautschnig, P. et al. CLUSTER guide RNAs enable precise and efficient RNA editing with endogenous ADAR enzymes in vivo. Nat Biotechnol (2022).
本课题组与合作者首次揭示基因组“无用”序列 稳定染色质三维结构及细胞稳态机制
2021.11.04人类基因组中的非编码序列在多种生物学过程中发挥着至关重要的作用,如非编码RNA、启动子、增强子和转座子等。作为在基因组中占比约98%的非编码区域,仍有大量是功能未知的,这些曾被认为是基因组中“垃圾”的区域,已被逐渐证实存在重要功能。基因组的三维结构会影响基因的转录调控或其他细胞生命活动,然而,除了研究较多的增强子-启动子成环所形成的三维结构在调控基因表达上的作用外,其他大量非编码序列在形成和维持正确三维染色质结构上是否具有潜在功能,在目前的研究中是被忽视的一个方向。之前有研究表明,改变非编码序列能够改变染色质结构,如切除一些拓扑结构相关结构域(TAD)的边界序列能够导致异常的基因表达,诱发疾病产生。除了TAD边界序列这一特殊类型,非编码序列在结构上的重要性目前还没有系统性的研究,尤其是一些与TAD无关、无任何表观遗传信号或功能元件注释的区域。
2021年11月3日,加州大学圣地亚哥分校(UCSD)王巍课题组与北京大学魏文胜课题组在Science Advances上联合发表了题为Noncoding loci without epigenomic signals can be essential for maintaining global chromatin organization and cell viability的研究论文,该研究基于分析得到的能够形成大量三维互作的非编码区域hub,通过CRISPR/Cas9系统对非编码hub位点进行靶向片段删除进而进行高通量功能性筛选,发现了一些不包含任何表观遗传标记的位点能够显著影响细胞生长和存活,并对于稳定全局染色质结构至关重要。
该研究基于来源于7个人类正常细胞系和癌细胞系中的5-kb分辨率的Hi-C数据(Rao et al. Cell 2014),将其中显著的染色体内互作对整合为片段互作网络,发现了其中与维持染色体三维结构至关重要的互作中心位点hub。这些位点聚集于异染色质区域,染色质开放性较低,且与已注释位点(包括蛋白编码基因和非编码RNA等)重合性较少(图1A-B)。为了探究hub位点是否对于稳定染色质结构具有重要作用,该研究基于实验室建立的CRISPR/Cas9系统介导的pgRNA片段删除策略(Zhu et al. Nature Biotechnology 2016),在K562细胞中对其中960个非编码hub位点进行高通量功能性筛选,发现了35个影响细胞存活或增殖的重要功能区(图1C)。其中,hub_22_7(chr22: 17,325,000-17,330,000, hg19)位点的切除能够在K562细胞系中导致显著的细胞死亡或细胞生长抑制,而不影响其他多种癌细胞(图1D)。

图1 hub位点的发现及高通量功能性筛选
筛选得到的hub位点不与任何蛋白编码基因、非编码RNA和TAD有重叠,其中77.1%(27/35)无任何组蛋白修饰或转录因子结合位点,包括hub_22_7位点(图2A)。相比于切除基因组安全位点AAVS1,Hi-C分析表明该位点的切除会在多个染色体上产生大范围染色质结构的变化。单细胞测序进一步发现,该hub位点的切除会激活一系列细胞凋亡相关基因的表达,改变同一染色体上远端区域多个与细胞存活相关基因如THOC5的表达,结合Hi-C数据发现其增强子与启动子的互作发生破坏(图2B-D)。由此表明,hub位点的破坏所诱导产生的染色质结构改变能够影响远端基因的转录调控,进而影响细胞功能。该研究首次揭示了无任何表观遗传信号的非编码位点能够稳定染色质三维结构并维持细胞存活,通过对这类hub位点的发现、筛选与功能解析,将进一步丰富对基因组非编码区域的功能阐述以及揭示其在维持三维基因组结构中的重要作用。
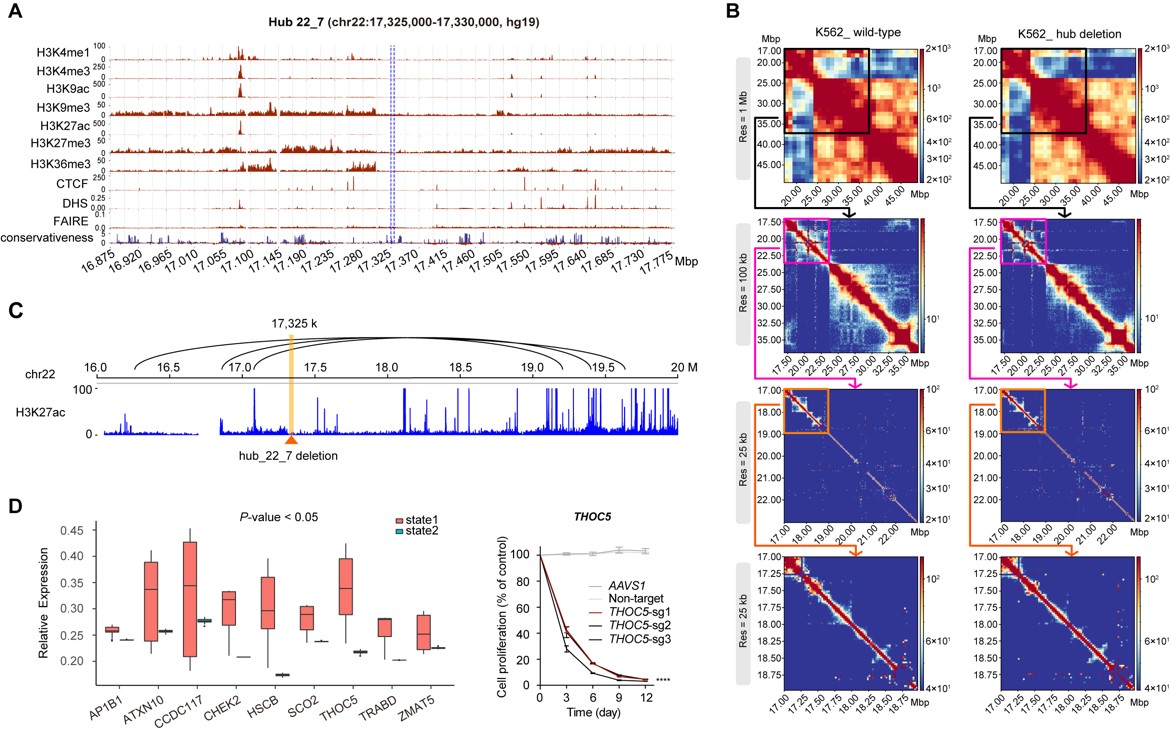
图2 hub位点切除导致染色质三维结构和基因表达的同时改变
加州大学圣地亚哥分校丁博博士,北京大学刘莹博士、刘志恒博士和UCSD博士生郑丽娜为该论文的共同第一作者,加州大学圣地亚哥分校王巍教授和北京大学魏文胜教授为论文共同通讯作者。该研究项目得到了加州再生医学研究所(CIRM)、美国国立卫生研究院(NIH)、国家自然科学基金、北京市科委生命科学前沿创新培育项目、北京未来基因诊断高精尖创新中心、北大-清华生命科学联合中心以及中国博士后科学基金的支持。
原文链接:https://www.science.org/doi/10.1126/sciadv.abi6020
本课题组和合作者报道新型冠状病毒激活宿主天然免疫反应新机制
2021.11.042021年11月3日,北京大学魏文胜课题组与中国医学科学院/北京协和医学院王健伟课题组合作在Signal Transduction and Targeted Therapy杂志发表了题为Sensing of cytoplasmic chromatin by cGAS activates innate immune response in SARS-CoV-2 infection的研究论文,报导了新冠病毒激活宿主天然免疫反应新机制。
由新冠病毒感染引起的2019年新型冠状病毒疾病(COVID-19)严重影响人类健康和社会稳定,但其致病机制仍不清。天然免疫反应是宿主抵抗病毒入侵的第一道防线,魏文胜课题组与合作者在前期发现,COVID-19病人肺部中存在强烈的干扰素活化特征[1],提示新冠病毒感染能够有效激活天然免疫活化途径。
新冠病毒是一类RNA病毒,通常由宿主RIG-I样受体(RLR)进行识别并激活干扰素反应。前期工作表明,新冠病毒在感染细胞的早期能够强烈抑制RLR信号通路;但在感染后期仍有大量干扰素产生[2,3],提示存在RLR非依赖的抗病毒信号传导途径的参与。该研究发现,新冠病毒感染能够诱导细胞融合形成大量的合胞体,在此过程中,细胞核中的染色质被运输到细胞质中,并被胞质中的DNA感受器cGAS识别,从而激活STING及下游通路,诱发宿主的抗病毒天然免疫反应。在新冠病毒感染小鼠的肺脏中,同样发现了合胞体的产生和STING通路的活化,提示该现象具有生理和病理意义。
为了进一步研究新冠病毒诱导的细胞融合与天然免疫活化的关系,课题组建立了由病毒刺突蛋白Spike及病毒受体ACE2相互作用所介导的细胞融合体系。结果表明,在非感染的条件下,由Spike-ACE2介导的细胞融合引起了细胞核机械力感知(Nuclear mechanosensing)的变化,导致核纤层和染色质稳定降低,从而促进了染色质的核输出(图1)。被运输到胞质中的染色质片段能够强烈激活cGAS-STING通路,促进干扰素的产生,并抑制新冠病毒的复制。该研究还发现,STING激动剂具有很强的抑制新冠病毒复制的功能,该结果为抗病毒治疗提供了新的思路。
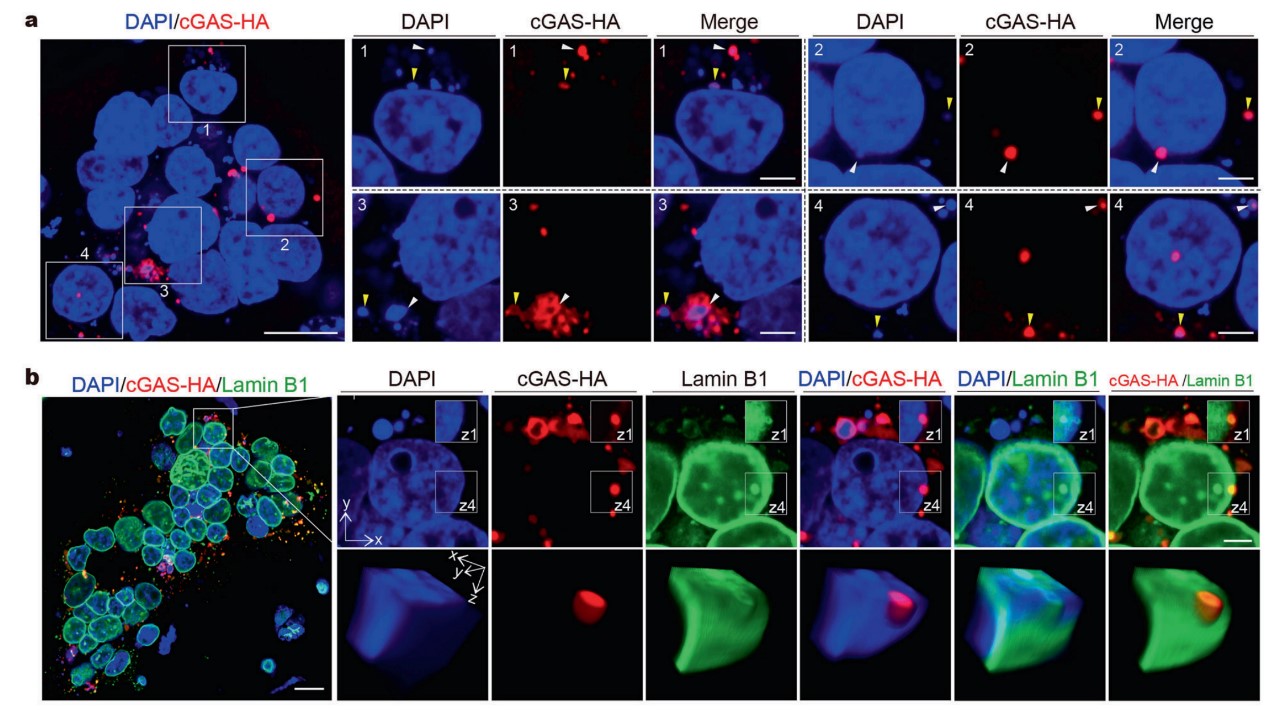
图1.细胞融合引起的染色质出核及cGAS对染色质片段的识别
天然免疫反应的经典范式是机体对“自我”和“非我”成分进行识别和区分后,针对病原体入侵做出的特异性反应。该研究发现,在一定情况下,宿主自身的成分可以被用作“危险信号”来激活抗病毒天然免疫反应。相关结果是对“病原相关分子模式”-“模式识别受体”这一理论的补充,并为理解新冠病毒与宿主相互作用机制提供了新的依据。
魏文胜课题组周卓副研究员为该论文的共同第一作者及共同通讯作者,课题组博士生张心怡以及中国医学科学院病原生物学研究所雷晓波研究员、肖霞博士为共同第一作者。中国医学科学院病原生物学研究所王健伟教授和魏文胜教授为本文的共同通讯作者。北京大学蒋争凡教授和哈佛大学医学院窦植洵教授对本文有重要贡献。该研究得到了国家重点研发计划、国家自然科学基金、传染病防治国家科技重大专项、中国医学科学院创新工程、北京市科委生命科学前沿创新培育项目、北京未来基因诊断高精尖创新中心等基金支持。
原文链接:https://www.nature.com/articles/s41392-021-00800-3
1. Zhou Z, Ren L, Zhang L, Zhong J, Xiao Y, Jia Z, Guo L, Yang J, Wang C, Jiang S, Yang D, Zhang G, Li H, Chen F, Xu Y, Chen M, Gao Z, Yang J, Dong J, Liu B, Zhang X, Wang W, He K, Jin Q, Li M, Wang J. Heightened innate immune responses in the respiratory tract of COVID-19 cases. Cell Host & Microbe. 2020, June 27(10),1-8.
2. Lei X, Dong X, Ma R, Wang W, Xiao X, Tian Z, Wang C, Wang Y, Li L, Ren L, Guo F, Zhao Z, Zhou Z, Xiang Z, Wang J. Activation and evasion of type I interferon responses by SARS-CoV-2. Nature Communications. 2020, 11(1), 3810.
3. Wang W, Zhou Z, Xiao X, Tian Z, Dong X, Wang C, Li L, Ren L, Lei X, Xiang Z, Wang J. SARS-CoV-2 nsp12 attenuates type I interferon production by inhibiting IRF3 nuclear translocation. Cellular & Molecular Immunology. 2021, Feb 18, 945–953
本课题组和合作者报道新冠病毒新型潜在受体发现
2021.08.20新型冠状病毒肺炎(COVID-19)的爆发已经在全球范围内引发了严重的公共卫生危机。SARS-CoV-2是导致COVID-19的主要病原体,它是继SARS-CoV和MERS-CoV之后第三种对人类具有高致病性的冠状病毒[1]。研究表明,ACE2(血管紧张素转化酶2)是介导SARS-CoV-2入胞的高亲和力受体,SARS-CoV-2通过其表面刺突蛋白(Spike,S)的受体结合域(RBD)与ACE2互作而感染细胞。然而,ACE2在人体器官中的表达分布与SARS-CoV-2的器官嗜性并不完全相关。比如,在整个呼吸道、大脑以及多种免疫细胞中,ACE2的表达较低或几乎不表达,但是这些细胞仍然可以被SARS-CoV-2感染。因此,可能存在ACE2以外的受体来介导病毒进入宿主细胞。
目前,已有多项研究发现与SARS-CoV-2感染相关的关键宿主因子,比如基于CRISPR基因敲除系统的高通量功能性筛选。然而,这些筛选均没有发现ACE2以外的新受体,这可能是因为此类基于功能缺失的筛选是在ACE2的表达和功能占主导地位的细胞类型中进行的[2-5]。
2021年8月20日,北京大学魏文胜课题组、中国医学科学院/北京协和医学院王健伟课题组与北京大学肖俊宇课题组在Science China Life Sciences杂志联合发表了题为Genome-wide CRISPR activation screen identifies candidate receptors for SARS-CoV-2 entry的研究论文,该研究通过基于CRISPR激活(CRISPR activation, CRISPRa)系统的全基因组水平的功能获得性筛选,发现了多个介导SARS-CoV-2入侵细胞的潜在新受体。
为了系统性研究SARS-CoV-2入胞的关键因子,该研究使用SARS-CoV-2假病毒在HEK293T细胞中进行了全基因组CRISPRa筛选。该筛选基于实验室之前建立的内置分子条形码(iBAR)方法[6]在sgRNA中添加外置条形码(eBAR),建立sgRNAeBAR文库,从而可以使用高感染复数及较少的细胞量进行高质量文库构建。筛选结果表明,除了发现已知的SARS-CoV-2受体ACE2、主要宿主细胞蛋白酶TMPRSS2以及已报道的ACE2依赖性共受体NRP1外,还鉴定出多种新型宿主因子可能参与SARS-CoV-2入胞机制中(图1)。
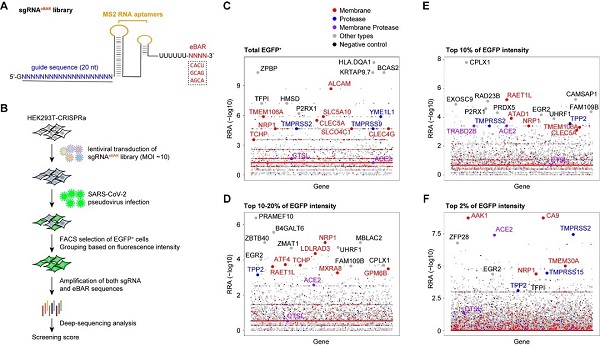
图1 通过全基因组CRISPRa功能性筛选发现影响SARS-CoV-2入胞的关键宿主因子
结合SARS-CoV-2假病毒和真病毒实验,该研究发现膜蛋白LDLRAD3、TMEM30A和CLEC4G能够以不依赖于ACE2的方式有效介导病毒入侵细胞。此外,研究证实这些膜蛋白都能够与SARS-CoV-2 S蛋白结合。与ACE2结合S蛋白的RBD不同,这些蛋白能够特异性结合S蛋白的N末端结构域(NTD)(图2)。LDLRAD3在神经元中高度表达,最近作为委内瑞拉马脑炎病毒(VEEV)的关键受体受到关注。实验表明,敲低LDLRAD3或在细胞上清中加入其可溶性蛋白可显著降低SARS-CoV-2对神经元细胞的感染。CLEC4G在肝脏、淋巴结和单核细胞中高度表达,已知其作为细胞粘附因子促进SARS-CoV感染宿主;通过在肝脏细胞中进行敲低实验证实了CLEC4G在SARS-CoV-2入侵细胞中的重要作用。跨膜蛋白TMEM30A在最近的SARS-CoV-2全基因组敲除筛选(Huh7.5细胞)中也被鉴定出来[4],研究进一步证实了其对于SARS-CoV-2入胞的重要性以及其与S蛋白的直接结合。这些具有组织特异性或广谱表达的新受体的发现,有助于进一步揭示SARS-CoV-2的多器官嗜性,并为探究COVID-19的新治疗靶点提供线索。

图2 SARS-CoV-2感染实验证实LDLRAD3、CLEC4G和TMEM30A为介导SARS-CoV-2入胞的潜在受体
北京大学魏文胜课题组博士后朱诗优博士、刘莹博士、副研究员周卓博士和肖俊宇课题组博士生张志莹为该论文的共同第一作者。该研究项目得到了国家重点研发计划、国家自然科学基金、传染病防治国家科技重大专项、北京市科委生命科学前沿创新培育项目、北京未来基因诊断高精尖创新中心、北大-清华生命科学联合中心以及中国博士后科学基金的支持。
原文链接:http://engine.scichina.com/doi/10.1007/s11427-021-1990-5
参考文献:
本课题组报道新型人源艰难梭菌毒素B受体
2021.08.13感染是世界范围内医源性感染的最主要原因之一1。它能够引起发热、腹泻、伪膜性肠炎、肠穿孔,甚至败血症、休克等危及生命的症状2。艰难梭菌通过分泌两种结构相似的毒素A和B(TcdA和TcdB)对宿主细胞造成伤害。梭菌毒素羧基端含有一段重复序列,被称为组合重复寡肽(combined repetitive oligopeptides, CROPs)。早期研究发现,CROPs蛋白能够竞争性抑制艰难梭菌毒素与细胞的结合,同时抗CROPs区域的抗体能够阻碍艰难梭菌毒素发挥细胞毒性3,4。因此,很长一段时间内人们认为宿主细胞表面受体是通过和艰难梭菌羧基端的CROPs区域相互作用介导毒素入胞。但随着CROPs缺失的梭菌毒素被发现,梭菌毒素入胞的分子机制被进一步完善5,6。梭菌毒素分别由CROPs依赖和非CROPs依赖的双受体介导入胞的模型得到了广泛的认同。随着基因编辑技术在艰难梭菌受体研究中的应用,硫酸软骨素蛋白聚糖4(CSPG4)、Frizzled-1/2/7等TcdB受体先后被报道7,8。然而,目前已知的TcdB受体主要都是通过与毒素非CROPs区域发生相互作用介导毒素内吞,而对于CROPs依赖的受体还不甚了解,阻碍了对其致病机制的进一步认知。
近日,本课题组在Science China Life Sciences 发表了题为Low-density lipoprotein receptor-related protein 1 is a CROPs associated receptor for Clostridioides difficile toxin B的研究论文。该研究发现低密度脂蛋白受体相关蛋白1(LRP1)是一种CROPs依赖的艰难梭菌毒素B受体。
在该研究中,研究人员基于CRISPR/Cas9技术,设计合成了靶向约3000个膜蛋白相关基因的亚基因组sgRNA文库。由于艰难梭菌毒素受体的冗余性为未知受体的发现带来了很大的困难。为降低TcdB受体冗余性对筛选造成的干扰,研究人员选择在CSPG4敲除细胞系中对未知的TcdB受体进行筛选。最终,在该研究中筛选得到了TcdB受体候选基因LRP1。后续研究人员通过基因敲除和基因上调等方法验证了LRP1介导艰难梭菌毒素入胞的生物学功能。有趣的是,与CSPG4和Frizzled-2不同,免疫共沉淀实验证明了LRP1与TcdB的相互作用仅需要通过CROPs即可完成。研究人员在CSPG4和FZD2双敲除的细胞系中敲除LRP1,进一步提高了细胞对于TcdB的耐受性,这也充分说明了三种受体功能上的冗余性。该研究通过发现CROPs依赖的TcdB受体证实了CROPs区域在介导TcdB与细胞膜表面受体中的作用,进一步完善了艰难梭菌毒素入胞分子机制,为艰难梭菌感染的治疗提供了潜在的靶点和理论依据。
北京大学生命科学学院郭生杰博士为该论文的第一作者,魏文胜教授和周卓副研究员为该论文的共同通讯作者,北京大学生命科学学院陈一欧博士、刘竞泽博士、博士研究生张心怡、刘志恒博士也参与了该研究工作。
文章链接:https://doi.org/10.1007/s11427-021-1943-9.
1 Martin, J. S. H., Monaghan, T. M. & Wilcox, M. H. Clostridium difficile infection: epidemiology, diagnosis and understanding transmission. Nat Rev Gastro Hepat 13, 206-216, doi:10.1038/nrgastro.2016.25 (2016).
2 Rupnik, M., Wilcox, M. H. & Gerding, D. N. Clostridium difficile infection: new developments in epidemiology and pathogenesis. Nature reviews. Microbiology 7, 526-536, doi:10.1038/nrmicro2164 (2009).
3 Frey, S. M. & Wilkins, T. D. Localization of two epitopes recognized by monoclonal antibody PCG-4 on Clostridium difficile toxin A. Infection and immunity 60, 2488-2492 (1992).
4 Sauerborn, M., Leukel, P. & von Eichel-Streiber, C. The C-terminal ligand-binding domain of Clostridium difficile toxin A (TcdA) abrogates TcdA-specific binding to cells and prevents mouse lethality. FEMS microbiology letters 155, 45-54, doi:Doi 10.1016/S0378-1097(97)00365-0 (1997).
6 Schorch, B. et al. LRP1 is a receptor for Clostridium perfringens TpeL toxin indicating a two-receptor model of clostridial glycosylating toxins. Proceedings of the National Academy of Sciences of the United States of America 111, 6431-6436, doi:10.1073/pnas.1323790111 (2014).
7 Yuan, P. et al. Chondroitin sulfate proteoglycan 4 functions as the cellular receptor for Clostridium difficile toxin B. Cell research 25, 157-168, doi:10.1038/cr.2014.169 (2015).
8 Tao, L. et al. Frizzled proteins are colonic epithelial receptors for C. difficile toxin B. Nature 538, 350-355, doi:10.1038/nature19799 (2016).
祝贺李齐恒、刘竞泽和许萍同学获得博士学位
2021.07.13祝贺李齐恒、刘竞泽和许萍同学分别于2020年12月和2021年6月顺利通过学位论文答辩,获得博士学位!
愿各位同学今后在新的工作岗位,向下根扎得更深,向上枝叶长得更好,为实现伟大的中国梦做出自己应有的贡献!祝各位同学毕业快乐,前程似锦!




祝贺本课题组张小雪同学和徐艺源同学获得2021-2022年度校长奖学金
2021.06.22根据《北京大学博士研究生校长奖学金管理办法》,经过前沿交叉学院和生命科学学院组织的答辩和评审工作,本实验室2017级博士研究生张小雪同学和2019级博士研究生徐艺源同学获得了2021-2022年度校长奖学金。向两位同学表示热烈祝贺!
张小雪同学和徐艺源同学在课程学习、学术研究等方面均有优异表现,相信在今后的学习和科研中,将更好的发挥示范带动作用,鼓励本实验室的同学们更加专注学业精进,实现品学兼优、全面发展。
本课题组报道基于碱基编辑的新型高通量功能性筛选方法
2021.06.212021年6月21日,魏文胜课题组在Nature Biotechnology杂志在线发表了题为 “Genome-wide interrogation of gene functions through base editor screens empowered by barcoded sgRNAs”的研究论文。
基于CRISPR-Cas的功能性筛选技术为基因功能研究和药物靶点发现提供了有力手段。经典的CRISPR敲除筛选方法依赖于Cas9介导的双链DNA断裂。然而,DNA双链断裂的产生如果修复不及时会造成严重的细胞损伤。因此,Cas9靶向基因组多拷贝位点时极易引起细胞死亡,从而影响基因功能筛选的准确性。此外,在对基因组损伤更为敏感的原代细胞中,基因编辑产生的DNA切割会激活p53信号通路,从而引起细胞周期停滞乃至细胞死亡,使得在细胞中进行大规模筛选比较困难。比如在有野生型p53表达的人视网膜色素上皮细胞中,经典CRISPR基因敲除筛选呈现出信噪比过低的结果;在人多能干细胞中,对单个基因组位点的编辑也同样容易造成细胞死亡。近期有多篇报道针对如何在原代细胞中实现大规模基因组功能性筛选的技术方案展开了争论和探讨,但始终没有令人满意的解决方案。
针对上述问题,魏文胜课题组发展了名为iBARed cytosine Base Editing-mediated gene KnockOut (BARBEKO)的新型筛选方案。该方法利用胞嘧啶单碱基编辑器,通过靶向破坏蛋白质编码基因的起始密码子位点或剪接位点、或通过引入提前终止密码子的方式来实现非依赖DNA双链断裂的高效基因敲除。通过结合实验室之前建立的内置分子条形码(iBAR)设计sgRNA (Zhu et al. Genome Biology 2019),BARBEKO方案可以依赖高病毒感染复数建库,在提升筛选效率的同时极大减少了所需细胞数量。相较于经典的CRISPR基因敲除筛选,BARBEKO方法能将筛选所需细胞量降低10-100倍,并能够通用于正向和负向选择筛选,可以大幅提升全基因组水平功能性筛选的可行性与经济性。
方法成功在多种癌细胞系和表达野生型p53的正常细胞中实现了准确、高效的细胞适应性(fitness)筛选。该方法能有效避免基因拷贝数效应,有效降低负向筛选中与细胞死活相关的高假阳性率;在对DNA双链断裂敏感的细胞系中,该方法在筛选精度和准确率方面优势尤为明显(图1)。这些结果表明,BARBEKO方法为原代细胞、类器官或模式动物体内等复杂生物模型的高通量筛选提供了更优的选择;为数量受限的细胞样品(如病人来源的细胞)提供了高效筛选方案,因此是基因功能和临床前研究的有力工具。

图1 | BARBEKO筛选策略有效提升基因敲除筛选效率和准确性
北京大学魏文胜课题组博士生许萍、刘志恒博士、刘莹博士和博士生马华峥为该论文共同第一作者。该研究项目得到了国家自然科学基金重点项目、北京市科委生命科学前沿创新培育项目、北京未来基因诊断高精尖创新中心和北大-清华生命科学联合中心的基金支持。
课题组全员前往香山远足
2021.06.17自2019年末新冠肺炎疫情爆发以来,课题组已经有一年半的时间没有集体出游了。在这个盛夏的毕业季,实验室精心组织了一场香山行,为忙于科研的大家放松减压,也为实验室的毕业生送上毕业祝福。
5月29日早8点30分,大家在香山公园东门准时集合,从山脚下的勤政殿出发,开始了这次的登山之行。前半段的山路较为平缓,大家有说有笑地缓步前进,也在“双清别墅”接受了爱国主义教育;后半段的山路渐渐陡峭,但是仍然没有阻挡大家向顶峰迈进的热情,虽然在夏日的骄阳下晒得有些汗流浃背,但是在半山腰稍作休息之后,大家继续迈开脚步,拾级而上,终于在10:30到达了香山的顶峰——香炉峰。

香山位于北京城西,这一天也是天朗气清,惠风和畅,站在山顶望去,整个北京城区尽收眼底。正所谓“凭高眺远,见长空,万里云无留迹”。大家在香炉峰合影留念,记录下这属于欢声笑语的美好时刻。

在我们每个人的人生路上都有一座又一座的山峰,它可以是眼前的课题实验,也可能是生活中的各种挑战。但是,我们每个人都要有“世上无难事,只要肯登攀”的精神,努力向前迈进,直到登上顶峰。在科研的大路上,我们Wei Lab也是一次又一次攀登科研高峰,并取得了不俗的成绩。祝福Wei Lab与Wei Lab的每位成员,都能在科研和人生上勇攀高峰,不断登顶!
供稿人:牛煦然
