新闻动态NEWS
您现在所在的位置: 首页» 新闻动态 NewsCell | 魏文胜团队与合作者联合开发新型通用型CAR-T 疗法治疗血液系统恶性肿瘤
2025.08.22过去十年间,自体CAR-T疗法在血液系统恶性肿瘤治疗中取得革命性突破。多款针对CD19和BCMA的产品已获批,在复发/难治性白血病、淋巴瘤和多发性骨髓瘤中显示出高缓解率,并为部分患者带来长期治愈,奠定了其作为精准免疫治疗“明星选手”的地位。
然而,自体CAR-T依赖患者自身T细胞,生产周期需 2–4 周且成本高昂。更重要的是,许多晚期患者因免疫系统严重受损,不仅缺乏可用于制备的健康T细胞,其体内微环境也不利于CAR-T的存活和扩增,从而失去治疗机会。基于此,“通用型CAR-T”(异体来源CAR-T)成为研究热点,即直接利用健康供体T细胞改造后输入患者,实现现货化和规模化生产。但在临床应用中,该策略需跨越多重免疫屏障:供体T细胞可能攻击患者组织,引发移植物抗宿主病(GvHD);而患者自身的T细胞和NK细胞也会迅速清除这些“外来者”,导致疗效难以持久。尽管基因编辑技术为解决这些难题提供了契机,但如何同时规避宿主T细胞和NK细胞的双重清除,仍是尚未攻克的核心瓶颈。
2025年8月21日,北京大学魏文胜团队、解放军总医院韩为东团队联合博雅辑因,在 Cell 发表题为“Glycan shielding enables TCR-sufficient allogeneic CAR-T therapy” 的研究论文。团队通过全基因组CRISPR筛选,鉴定出糖基化调控基因SPPL3,并提出“糖盾护体”策略。研究显示,在供体T细胞中敲除SPPL3并导入抗 CD19 CAR,可在细胞表面形成致密糖基化修饰层,从而通过三重机制实现免疫保护:其一,降低HLA-I分子暴露,削弱宿主T细胞的识别;其二,下调NK细胞活化配体,增强抵御宿主NK细胞杀伤的能力;其三,抑制Fas/FasL途径介导的细胞死亡。值得强调的是,这一改造并未影响 CAR-T 的抗肿瘤效能。体外实验和动物模型均证实,SPPL3缺失的CAR-T在清除靶肿瘤方面与对照组表现相当,却显著提升了免疫耐受性和持久性。
为寻找能够同时规避宿主与CAR-T细胞免疫互斥的工程化策略,魏文胜团队利用全基因组 CRISPR 敲除文库,系统筛选影响异体T细胞持久性的关键基因及其调控网络。研究设想通过筛选获得三类功能改造:(1)降低HLA-ABC的可检测性,从而削弱TCR介导的识别与清除;(2)增强对NK细胞杀伤的抵抗力;(3)提升长期存活性,尤其是抵御FasL诱导的凋亡。令人意外的是,这三类表型最终均指向同一关键基因——SPPL3(图一)。其敲除不仅可同时降低HLA-ABC的表面可及性、增强抗NK细胞能力并延长T细胞存活,还揭示了一个保守而普遍的免疫调控机制。已有研究为此提供了重要线索:肿瘤细胞缺失 SPPL3后可借此机制逃避免疫清除1;韩为东团队在肿瘤抵抗CAR-T杀伤的全基因组筛选中同样发现SPPL3排名首位2;Singh团队则报道Nalm6肿瘤细胞在SPPL3缺失后获得对CAR-T杀伤的抵抗3。这些发现共同提示,SPPL3在癌症免疫逃逸与T细胞免疫耐受中具有普遍作用。

图一、多重筛选锚定异体T细胞存活的关键靶点SPPL3
SPPL3 编码一种定位于高尔基体的膜内切割酶,可通过抑制多种糖基转移酶和糖苷酶的活性,从而负向调控细胞表面糖链的合成。研究发现,敲除 SPPL3 能显著提高T细胞表面的整体糖基化水平,形成致密的糖基修饰层,即所谓的“糖苷盾”。这一“护盾”在多个免疫识别环节中发挥保护作用:其一,降低HLA-I分子的表面可及性,从而削弱了宿主T细胞通过TCR介导的识别与清除;其二,减少NK细胞活化配体的暴露,增强对宿主NK细胞杀伤的抵御能力;其三,降低Fas分子的表面可及性,抑制Fas/FasL途径触发的活化诱导性细胞死亡(AICD)。
更为关键的是,SPPL3的基因编辑并未损害 CAR-T 的肿瘤杀伤效能。无论在体外细胞毒性实验,还是在动物模型验证中,SPPL3 缺失的CAR-T清除靶肿瘤的效率均与未编辑的对照相当。这表明,“糖苷盾”策略不仅显著提升了免疫耐受性和持久性,同时仍保留CAR分子介导的强大抗肿瘤活性。
研究团队先后开展了两项IIT临床试验。第一项试验采用TCR与SPPL3双敲除策略,结果显示在安全性方面未出现 GvHD,但 CAR-T 在体内的持续时间有限(图二)。

图二、TCR与SPPL3双敲除策略的CAR-T的临床结果
通过对输入产品中残存CD3+ CAR-T的扩增动力学分析,研究者推测SPPL3敲除所形成的“糖苷盾”能够避免GvHD,而保留TCR基底信号则可能对维持T细胞长期存活至关重要。基于这一假设,团队进一步开展了仅敲除SPPL3、保留TCR的第二项临床试验。结果显示,患者体内的CAR-T可持续存在6个月以上,未观察到典型GvHD或严重免疫并发症,部分患者获得持久缓解(图三)。
这一结果首次证明:SPPL3缺失诱导的糖基化重塑能够突破异体CAR-T的免疫屏障,而内源性TCR信号对细胞持久性具有不可替代的作用。该发现为通用型CAR-T的设计与开发提供了全新的策略范式。

图三、SPPL3单敲策略的CAR-T的临床结果
在临床观察中,研究团队首次提出并界定了“双向同种异体T细胞反应综合征”(BATCRS)。该综合征描述了在异体环境下,供体与受体T细胞同时发生的轻度双向免疫应答,其表现为局部炎症反应,但并不导致病理性组织损伤,具有自限性,仅需支持治疗即可缓解。其独特的免疫学特征确立了BATCRS作为独立综合征的地位,为优化异体细胞治疗的安全性与有效性提供了新的临床依据。
本研究通过全基因组筛选提出了突破通用型CAR-T疗法免疫屏障的全新策略。SPPL3靶向编辑不仅保留了CAR介导的抗肿瘤效能,还显著增强了免疫耐受性与体内持久性,提供了一种能够保留TCR的通用型CAR-T可行方案(图四)。这一发现不仅拓展了通用型免疫细胞疗法的设计思路,也为γδT细胞和NK细胞等多种通用型细胞治疗提供了潜在的分子改造靶点。随着机制研究深入和更大规模临床验证的开展,“糖盾护体”策略有望成为异体免疫细胞疗法的重要模块化工具,加速“现成即用”免疫治疗在更多癌种中的转化与应用。

图四、SPPL3敲除助力异体CAR-T细胞免疫逃逸并提升持久性
本研究通讯作者为北京大学魏文胜教授,中国人民解放军总医院韩为东教授,以及博雅辑因袁鹏飞博士。共同第一作者包括武泽光博士、施锦红(博士研究生)、拉毛切忠博士、邱圆圆(博士研究生)、杨金鑫(博士研究生)、刘洋副主任医师和博雅辑因梁霏霏博士。质谱分析工作得到了黄超兰团队的大力支持。研究同时获得国家自然科学基金A+B的专项资助、科技部重点研发计划、重大慢性非传染性疾病科技专项、中国医学科学院医学与健康科技创新基金、人畜共患传染病重症诊治全国重点实验室开放课题、中国医学科学院人才计划以及北大-清华生命科学联合中心的资助。
原文链接:https://www.cell.com/cell/fulltext/S0092-8674(25)00910-9
参考文献
1. Jongsma, M.L.M., de Waard, A.A., Raaben, M., Zhang, T., Cabukusta, B., Platzer, R., Blomen, V.A., Xagara, A., Verkerk, T., Bliss, S., et al. (2021). The SPPL3-Defined Glycosphingolipid Repertoire Orchestrates HLA Class I-Mediated Immune Responses. Immunity 54, 132-150.e9. https://doi.org/10.1016/j.immuni.2020.11.003.
2. Yan, X., Chen, D., Wang, Y., Guo, Y., Tong, C., Wei, J., Zhang, Y., Wu, Z., and Han, W. (2022). Identification of NOXA as a pivotal regulator of resistance to CAR T-cell therapy in B-cell malignancies. Signal Transduct. Target. Ther. 7, 98. https://doi.org/10.1038/s41392-022-00915-1.
3. Heard, A., Landmann, J.H., Hansen, A.R., Papadopolou, A., Hsu, Y.-S., Selli, M.E., Warrington, J.M., Lattin, J., Chang, J., Ha, H., et al. (2022). Antigen glycosylation regulates efficacy of CAR T cells targeting CD19. Nat. Commun. 13, 3367. https://doi.org/10.1038/s41467-022-31035-7.
Nature Communications | 成环有道:魏文胜团队开发RNA环化新方法
2025.08.12环状RNA因其高度稳定的结构与高效蛋白编码能力,在RNA疗法领域备受关注。近年来,基于环状RNA的新型治疗策略已在疫苗、体内原位CAR-T、基因编辑及蛋白替代等方面取得进展。其中,魏文胜团队此前已报道多项环状RNA平台的重要应用成果,包括:环状RNA新冠疫苗(Cell,2022)1、利用RNA环化策略优化的RNA编辑工具LEAPER 2.0(Nature Biotechnology,2022)2,以及升级版线粒体基因编辑工具mitoBE v2(Nature,2025)3,在蛋白编码与非编码功能实现方面均展示了环状RNA平台的独特优势。
在环状RNA平台中,RNA的体外环化是实现其功能的关键环节。目前常用方法包括化学合成与连接法、酶连接法以及基于核酶(Ribozyme)的环化方法。其中,核酶方法因不依赖蛋白而具有更强的通用性,受到广泛关注。然而,这些方法仍存在局限:例如,应用最广的PIE(Permuted Intron-Exon)方法4需要对自剪接核酶(一型与二型内含子)进行拆分和改造,限制了可用内含子的来源与种类;此外,RNA环化的长度亦受制约;同时,环状RNA的潜在免疫原性以及纯化过程的复杂性,也成为其推广的障碍。这些挑战凸显了开发高效、普适且易用的新型体外环化技术的迫切需求。
2025年8月10日,北京大学/昌平实验室魏文胜团队在Nature Communications发表题为 “Self-splicing RNA circularization facilitated by intact group I and II introns” 的研究论文。团队基于一型与二型内含子的第二步剪接反应,开发了两种全新的体外RNA环化方法:PIET(Permuted Intron-Exon through Trans-splicing)与CIRC(Complete self-splicing Intron for RNA Circularization)。PIET是一种两组分RNA环化体系,可通过调节组分比例控制环化效率;而CIRC完全摒弃了内含子拆分与改造的需求,可直接利用多种天然完整内含子完成环化,显著拓宽了可选内含子范围,并为筛选更高效或具特殊功能的天然内含子提供了可能。与PIE相比,CIRC不仅环化效率更高,还能在低Mg²⁺或低pH等温和条件下保持高活性,有利于RNA完整性和质量保持。
在本研究中,CIRC方法突破了现有环状RNA平台的载量上限,首次成功环化12,206 nt、编码427 kDa全长抗肌萎缩蛋白(dystrophin)的RNA,并在细胞中实现了完整蛋白的表达。这一成果刷新了环状RNA可承载RNA分子的长度纪录,为杜氏肌营养不良症(DMD)等大蛋白缺陷相关疾病的蛋白替代疗法开辟了新路径。此外,CIRC方法还能实现无痕(scarless)环状RNA制备,产物免疫原性低,并可在内含子中引入poly(A)序列,从而与基于oligo(dT)的纯化体系兼容,大幅简化了生产纯化流程。
这一研究构想源于团队在2023年初使用PIE方法制备环状RNA时提出的假设:是否可跳过第一步剪接反应,直接利用第二步反应实现环化?实验验证了这一设想,并据此开发出依赖双组分RNA反式剪接活性的环化方法PIET(图1)。

图1 PIET方法的开发示意图
研究团队进一步推测,将PIET方法中的非共价连接改为共价连接,内含子依然可能保持活性。实验结果证实了这一推测,并发现去除PIE与PIET方法中所需的同源臂后,环化效率显著提高。由于该策略允许使用天然完整内含子实现RNA环化,研究者将其命名为CIRC(图2)。

图2 从PIET到CIRC
CIRC方法不仅能高效兼容原PIE方法中的内含子,还可适配十余种来自自然界的一型和二型内含子,证明了其广泛适用性。这为未来从自然界筛选更高效或具特定功能的内含子提供了更多可能。此外,与PIE方法相比,CIRC方法在温和条件下表现出更高的环化效率与反应速率,对保持长RNA的完整性尤为有利。利用这一优势,团队系统评估了CIRC方法在大分子RNA环化中的能力,成功制备载荷长度从1,448 nt到12,206 nt的环状RNA,并首次验证最大载荷RNA在细胞内的蛋白表达(图3),实现了全长抗肌萎缩蛋白的环化与表达,为相关疾病的大分子蛋白替代疗法奠定了技术基础。

图3 CIRC方法突破了目前环状RNA平台的载量限制
本研究不仅为RNA体外环化提供了全新的技术路线,也极大拓展了环状RNA平台的应用潜力,对未来RNA疗法的开发与优化具有重要意义。
魏文胜课题组前沿交叉学科研究院2020级毕业生沈勇博士与生命科学学院2024级博士研究生李帛翰为本论文共同第一作者。昌平实验室董蕾,北京大学前沿交叉学科研究院博士研究生唐玮(2021级)、任纪武(2023级)、陈奉(2021级)、生命科学学院2019级毕业生郑文娟博士、科研助理于莹博士与圆因生物高璐博士对本研究做出了重要贡献。研究获得了昌平实验室及北大-清华生命科学联合中心的资助。
文章链接:https://doi.org/10.1038/s41467-025-62607-y
参考文献:
1. Qu, L. et al. Circular RNA vaccines against SARS-CoV-2 and emerging variants. Cell 185, 1728-1744 e1716 (2022).
2. Yi, Z. et al. Engineered circular ADAR-recruiting RNAs increase the efficiency and fidelity of RNA editing in vitro and in vivo. Nat Biotechnol 40, 946-955 (2022).
3. Zhang, X. et al. Precise modelling of mitochondrial diseases using optimized mitoBEs. Nature 639, 735-745 (2025).
4. Wesselhoeft, R. A., Kowalski, P. S. & Anderson, D. G. Engineering circular RNA for potent and stable translation in eukaryotic cells. Nat Commun 9, 2629 (2018).
祝Wei Lab 2025届各位毕业生们毕业快乐
2025.07.032025年盛夏,Wei Lab迎来硕果累累的毕业季。实验室14位优秀学子圆满完成学业,其中徐艺源、郑文娟、陈盎、唐慧贤、牛煦然、张巍、沈勇7位同学获得博士学位;莫滨瑞、冯婉滢、杨悦、刘宙达、刘雨涵、谢雨萱、李小雨7位同学获得学士学位。

特别值得庆贺的是:
- 唐慧贤同学获得2025年“北京市普通高等学校优秀毕业生”及“北京大学优秀毕业生”双项殊荣;
- 牛煦然、沈勇同学获评2025年“北京大学优秀毕业生”称号;
- 牛煦然同学同时获得生命科学联合中心(北大方面)“生命之星”奖和北京大学生命科学学院优秀毕业研究生张景钺奖;
- 莫滨瑞同学的毕业论文获评2025年度北京大学优秀本科毕业论文;
- 冯婉滢同学的毕业论文获评2025年度北京大学生命科学学院优秀本科毕业论文。
让我们祝贺各位毕业生!
毕业是新征程的起点,衷心祝愿各位毕业生秉承北大精神,在未来的科研道路和人生旅途中勇攀高峰,续写精彩华章!Wei Lab永远是你们温暖的家,期待捷报频传!


Nature Biotechnology | 于无声处:魏文胜团队系统揭秘人类基因组中“沉默突变”的功能性“低语”
2025.06.25同义突变指DNA序列发生改变却未引起蛋白质氨基酸序列变更的突变,因而在传统遗传学理论中通常被视为"中性"1。然而近年来多项研究表明,这类"沉默突变"在病毒和原核生物中可能影响其适应度,提示其存在潜在的生物学功能2–5。特别是最近在酿酒酵母中的研究更是提出,同义突变与非同义突变同样显著地改变细胞适应度6,不过该研究结论存在较大争议7。尤其在人类等多细胞生物中,同义突变是否具备功能效应仍缺乏系统性实验证据。
2025年6月24日,北京大学/昌平实验室魏文胜团队在Nature Biotechnology杂志发表了题为“Prime editor-based high-throughput screening reveals functional synonymous mutations in human cells”的研究论文。该研究首次系统性揭示,同义突变虽整体上趋于中性,但部分突变仍具有重要生物学功能;同时,研究团队还提出了一套适用于精准突变研究的创新实验方法,为人类遗传学研究提供了新工具。
本研究基于PEmax基因编辑系统,构建了一个覆盖3,644个人类蛋白编码基因、共计297,900条epegRNA的突变文库,并在人类结肠癌细胞系HCT116中开展了迄今最为系统的同义突变高通量功能筛选。结果共鉴定出409个显著影响细胞增殖的同义突变,占全部同义突变的0.43%,显著低于1,505个具有功能效应的非同义突变(占全部非同义突变的3.83%)。整体分析显示,同义突变对细胞适应度的影响相对较弱,大多数呈中性状态,且与错义、无义和移码突变等非同义突变相比,其功能性差异显著(图1)。这一结果与此前在酿酒酵母中的相关研究结果明显不同。

图1基于高通量筛选策略在人类基因组中鉴定功能性同义突变
尽管大多数同义突变不具显著功能效应,研究仍识别出一小部分可能对细胞产生不利影响的同义突变。为探究其潜在机制,研究团队开发了机器学习模型DS Finder(图2),发现这些功能性同义突变通常通过破坏RNA剪接位点、改变mRNA二级结构或干扰翻译起始,进而影响基因表达与蛋白质合成。例如,突变PLK1_S2(AGT>AGC)可增强mRNA局部稳定性却抑制翻译起始;BUB1B_R322(AGG>AGA)则引发异常剪接,导致mRNA降解。

图2 应用机器学习及单细胞测序等多种方法探究人类基因组中的功能性同义突变
研究进一步整合筛选数据与DS Finder模型,对临床数据库中的同义突变开展系统性预测分析,成功识别出若干具有潜在致病意义的突变。其中,G6PC3 c.G399A此前被注释为“可能良性”,本研究首次证实其具有明确的功能效应,提示其在特定条件下可能参与疾病发生与发展(图3)。

图3 使用DS Finder在临床数据中预测潜在的功能性同义突变
本研究拓展了对“沉默突变”生物学功能的认知,并为绘制人类基因功能变异图谱提供了方法学上的突破。研究构建的突变筛选平台与预测模型已面向公众开放,欢迎访问项目网站“Hearing Silence”获取更多信息与使用工具(https://search-synonymous-mutations.streamlit.app/)。
北京大学生命科学学院博士研究生牛煦然、前沿交叉学科研究院博士研究生唐玮,以及出站博士后刘永烁博士(现任山东省肿瘤医院检验科副研究员)为本论文的共同第一作者,昌平实验室副研究员刘莹博士为共同通讯作者。北京大学生命科学学院本科生莫滨瑞与科研助理于莹博士亦为本研究做出重要贡献。项目得到了国家自然科学基金、北大-清华生命科学联合中心、昌平实验室以及泰山学者基金的支持。
文章链接:https://www.nature.com/articles/s41587-025-02710-z
参考文献:
1. Kimura, M. Preponderance of synonymous changes as evidence for the neutral theory of molecular evolution. Nature 267, 275–276 (1977).
2. Cuevas, J. M., Domingo-Calap, P. & Sanjuan, R. The fitness effects of synonymous mutations in DNA and RNA viruses. Mol. Biol. Evol. 29, 17–20 (2012).
3. Kristofich, J. et al. Synonymous mutations make dramatic contributions to fitness when growth is limited by a weak-link enzyme. PLoS Genet. 14, e1007615 (2018).
4. Walsh, I. M., Bowman, M. A., Soto Santarriaga, I. F., Rodriguez, A. & Clark, P. L. Synonymous codon substitutions perturb cotranslational protein folding in vivo and impair cell fitness. Proc. Natl. Acad. Sci. U S A 117, 3528–3534 (2020).
5. Lebeuf-Taylor, E., McCloskey, N., Bailey, S. F., Hinz, A. & Kassen, R. The distribution of fitness effects among synonymous mutations in a gene under directional selection. eLife 8, e45952 (2019).
6. Shen, X., Song, S., Li, C. & Zhang, J. Synonymous mutations in representative yeast genes are mostly strongly non-neutral. Nature 606, 725–731 (2022).
7. Kruglyak, L. et al. Insufficient evidence for non-neutrality of synonymous mutations. Nature 616, E8–E9 (2023).
祝贺Wei Lab多位毕业生获评荣誉称号和奖励奖学金
2025.06.09根据《北京大学学生奖励条例》及相关规定,按照《关于评选2025年“北京大学优秀毕业生”(夏季)和“北京市普通高等学校优秀毕业生”(夏季)的通知》相关要求,结合毕业生的在校表现、综合班级民主评议结果、答辩评审及推荐意见,经过生命科学学院和前沿交叉学科研究院奖励奖学金小组评审通过,唐慧贤同学(生命科学学院)获评2025年“北京市普通高等学校优秀毕业生”和“北京大学优秀毕业生”称号,牛煦然同学(生命科学学院)和沈勇同学(前沿交叉学科研究院)获评2025年“北京大学优秀毕业生”称号。
此外,经过个人申请、材料初筛、专业推荐和终审答辩,牛煦然同学获得了2025年生命科学联合中心(北大方面)“生命之星”奖(北京大学从事与生命科学领域相关研究的博士研究生申请,全校共评选五位获奖者)和2025年北京大学生命科学学院优秀毕业研究生张景钺奖(北京大学生命科学学院授予研究生毕业生的最高荣誉)。
唐慧贤同学、牛煦然同学和沈勇同学通过自己的勤奋努力,在与同组同学的合作下,均在多个课题研究领域获得了突破。辛苦付出终于获得回报,让我们祝贺三位优秀毕业生!
生命之星奖介绍:
生命科学联合中心是为了加速我国一流大学的建设,由国家支持,教育部、科技部、财政部联合组织设计,北京大学与清华大学密切配合,于2011年4月成立的人才培养与教育科研体制改革试点单位(简称“生命中心”)。为激励生命科学领域的博士研究生,生命中心北大方面特设立“生命之星”奖学金,面向北京大学从事与生命科学领域相关的博士研究生开放申请,资助优秀研究生,提高科研能力和创造力。
张景钺奖介绍:
张景钺先生,植物形态学家,教育家。
我国植物形态学和植物系统学的开拓者。自1932年起,长期担任北京大学生物系教授、系主任,为北大生物系最早一批研究生导师,培养了大批生物学人才,为中国高等学校生物学教育做出了重要贡献。
2018年5月,时任东宝实业集团有限公司董事长、通化东宝药业股份有限公司董事长、学院1970级校友李一奎,捐赠学院一千万元,设立北京大学生命科学学院东宝教育基金,助力学院教育事业和人才培养。基金项目包含“生命科学学院优秀毕业研究生张景钺奖”。
STTT | 魏文胜团队实现人类肿瘤免疫调控网络的单碱基精度解析
2025.03.20抗PD-L1/HLA-I免疫检查点阻断(ICB)疗法虽然革新了癌症的临床治疗模式,但肿瘤患者体内PD-L1或HLA-I的异常表达仍然显著影响其疗效1。近年来,多项研究利用CRISPR/Cas9筛选技术系统性揭示了调控肿瘤细胞PD-L1和HLA-I表达的关键因子2-5。然而,受传统CRISPR筛选体系分辨率的限制,目前研究主要集中在基因层面的调控机制解析,而对位点层面的精细调控网络仍缺乏深入理解。值得注意的是,肿瘤基因组中的体细胞突变可以通过调控这些关键免疫因子的表达,显著影响肿瘤的发生、发展及ICB疗法的敏感性。国际癌症基因组联盟(ICGC)数据库分析表明,单核苷酸变异(SNV)占体细胞突变的90%以上。然而,当前对这些遗传变异的生物学功能及其临床意义仍缺乏系统性认知,许多临床相关突变的功能尚未得到充分解析。

2025年3月19日,北京大学/昌平实验室魏文胜课题组在《Signal Transduction and Targeted Therapy》杂志发表了题为“Massively parallel interrogation of human functional variants modulating cancer immunosurveillance”的研究论文。该研究基于单碱基分辨率的高通量筛选技术,首次系统性鉴定出大量影响PD-L1和HLA-I表达的关键功能位点,构建了全新的肿瘤免疫调控分子图谱,为精准免疫治疗提供了重要理论基础。
课题组此前构建了一套基于腺嘌呤碱基编辑器(ABEmax)的sgRNA文库,包含约82万个sgRNA,覆盖人类基因组中所有可设计的潜在磷酸化修饰位点,包括丝氨酸、苏氨酸和酪氨酸6。本研究利用该sgRNA文库,在人恶性黑色素瘤细胞A375中进行高通量功能筛选(图1a),成功鉴定出上千个调控PD-L1和HLA-I表达的全新功能位点(图1b-d)。通过大规模单独验证,研究证实这些位点不仅包括已知基因上的未被报道的调控位点,还涵盖了一系列全新的调控基因及其关键功能位点,其中许多与磷酸化修饰密切相关,尤其是IFNγ信号通路相关的基因。
深入分析显示,这些突变的影响不仅限于磷酸化修饰过程的改变,还可通过多种机制调控基因功能,包括干扰mRNA或蛋白质稳定性、影响DNA结合能力、改变蛋白质-蛋白质相互作用以及调控酶活性等。这些多层次的调控作用最终影响免疫分子的表达水平,为解析肿瘤免疫微环境的动态调控提供了新视角。
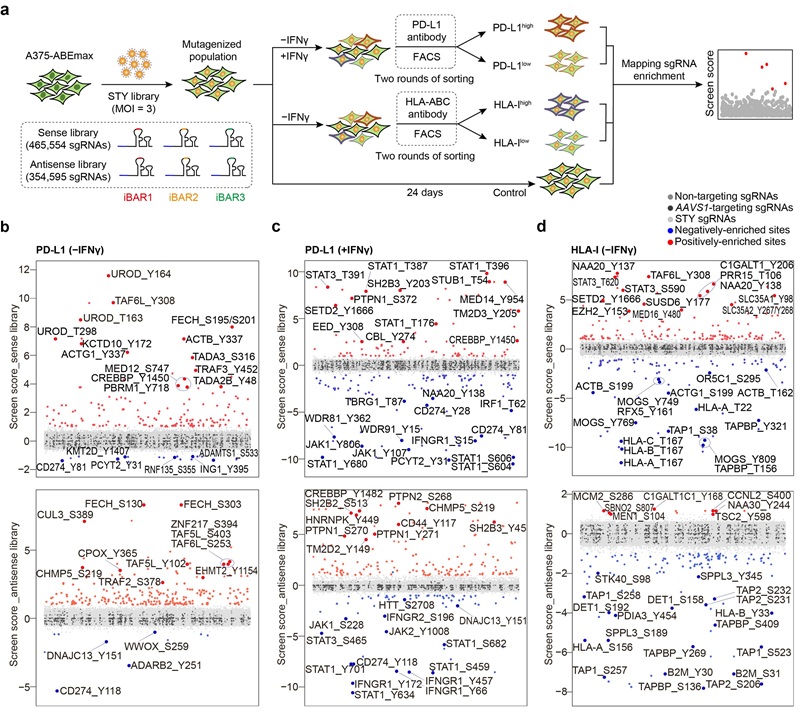
图1 基于ABEmax系统的高通量筛选鉴定调控PD-L1和HLA-I表达的功能位点
研究还发现了多种同时调控PD-L1和HLA-I表达的关键突变,例如临床相关突变SETD2_Y1666。SETD2是主要的组蛋白甲基转移酶,负责催化H3K36三甲基化(H3K36me3)。研究发现Y1666位点的突变虽不影响SETD2的mRNA和蛋白表达水平,却能严重破坏其H3K36me3的催化功能,进而导致PD-L1和HLA-I的同步上调。这一发现表明SETD2_Y1666突变可能通过调控染色质状态影响肿瘤免疫逃逸。
进一步的动物实验表明,在黑色素瘤和结肠癌模型中,该突变能够显著增强CD8+ T细胞的浸润及细胞毒性,并有效提高抗PD-1治疗的疗效(图2a–b)。此外,在多种人类肿瘤细胞系(黑色素瘤A375和A875、纤维肉瘤HT1080、乳腺癌MCF-7)中,该突变均表现出一致的免疫调控作用,凸显了其在临床应用中的广谱价值(图2c)。这一发现与近期多项临床研究结果相呼应,这些临床研究表明,携带SETD2失活突变的不同癌症类型的患者对ICB治疗具有更显著的响应。
本研究进一步整合ICGC和COSMIC数据库的多组癌症患者数据,发现本研究筛选出的关键位点在临床上具有较高的相关性,约40%的位点可在现有临床数据库中找到对应突变(图2d)。这一研究首次在单氨基酸/单碱基分辨率层面系统性解析了PD-L1和HLA-I的表达调控网络,为预测肿瘤免疫应答和ICB治疗效果提供了重要的临床生物标志物,同时也为癌症临床诊断、ICB治疗及创新药物研发提供了全新的视角。
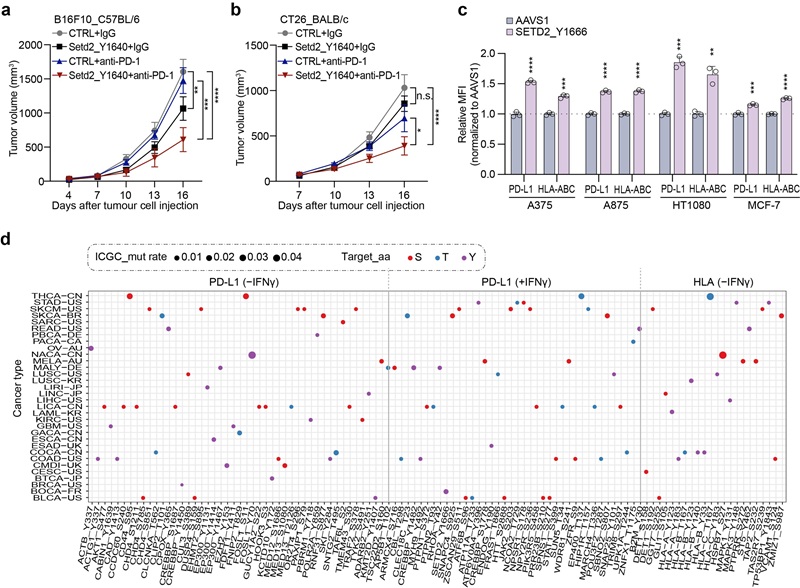
图2 筛选鉴定出的代表性临床相关位点及其在多种肿瘤模型和细胞系中的免疫调控功能
昌平实验室副研究员刘莹博士、北京大学已出站博士后刘永烁博士(现任山东省肿瘤医院检验科副研究员)、北京大学生命科学学院博士研究生牛煦然、前沿交叉学科研究院博士研究生陈盎以及昌平实验室博士后李依舟博士为该论文的共同第一作者。该研究得到了昌平实验室、国家自然科学基金、北大-清华生命科学联合中心、泰山学者基金及中国博士后基金的支持。
文章链接:https://www.nature.com/articles/s41392-025-02171-5
参考文献:
1 Sharma, P., Hu-Lieskovan, S., Wargo, J. A. & Ribas, A. Primary, Adaptive, and Acquired Resistance to Cancer Immunotherapy. Cell 168, 707-723, doi:10.1016/j.cell.2017.01.017 (2017).
2 Mezzadra, R. et al. Identification of CMTM6 and CMTM4 as PD-L1 protein regulators. Nature 549, 106-110, doi:10.1038/nature23669 (2017).
3 Suresh, S. et al. eIF5B drives integrated stress response-dependent translation of PD-L1 in lung cancer. Nat Cancer 1, 533-545, doi:10.1038/s43018-020-0056-0 (2020).
4 Dersh, D. et al. Genome-wide Screens Identify Lineage- and Tumor-Specific Genes Modulating MHC-I- and MHC-II-Restricted Immunosurveillance of Human Lymphomas. Immunity 54, 116-131 e110, doi:10.1016/j.immuni.2020.11.002 (2021).
5 Gu, S. S. et al. Therapeutically Increasing MHC-I Expression Potentiates Immune Checkpoint Blockade. Cancer Discov 11, 1524-1541, doi:10.1158/2159-8290.CD-20-0812 (2021).
6 Li, Y. et al. Functional profiling of serine, threonine and tyrosine sites. Nat Chem Biol, doi:10.1038/s41589-024-01731-0 (2024).
Nature | 魏文胜团队发布新一代线粒体碱基编辑器助力建立疾病动物模型
2025.01.20核基因组突变是多种疾病的根源,而线粒体作为细胞内具有半自主功能的细胞器,拥有独立的基因组,其基因组突变同样与多种遗传疾病密切相关。线粒体疾病通常累及多种组织器官,其中最为人熟知的包括Leigh综合征和LHON(Leber遗传性视神经病变)。Leigh综合征的症状包括发育迟缓、肌张力减退、运动和呼吸障碍等,而LHON则表现为视力丧失、中央暗点和视神经萎缩等问题。根据MITOMAP的统计,目前已验证的线粒体致病性突变有97个,其中点突变占比高达95%。然而,由于缺乏有效的点突变相关线粒体疾病小鼠模型,线粒体疾病的研究与治疗开发受到了严重制约。
早期的小鼠模型主要通过化学诱导或遗传工程构建[1],但这些方法操作复杂、成本高昂且对突变的精准控制较差,仅成功建立了极少数模型。近年来,研究人员成功开发了线粒体碱基编辑工具,可以对线粒体DNA实现C到T和A到G的编辑,例如DdCBEs和TALEDs。这些工具基于双链DNA脱氨酶DddA蛋白[2, 3]。虽然已有研究者尝试将这些工具应用于小鼠模型的构建,但其编辑效率尚不足以模拟人类线粒体疾病中高突变负荷的特征[4, 5]。此外,研究表明DdCBEs可能引发大量核基因组脱靶效应,这种非TALE依赖性的脱靶主要源于DddA蛋白的自组装以及其与CTCF的相互作用[6]。因此,基于DddA的线粒体碱基编辑工具在应用中面临核基因组脱靶的风险,难以直接建立线粒体突变与疾病表型之间的因果联系。
针对这一挑战,北京大学魏文胜课题组之前开发了mitoBEs,这是一种结合切口酶与单链DNA脱氨酶的新型线粒体碱基编辑工具,能够实现线粒体DNA的C到T和A到G编辑。与DdCBEs和TALEDs相比,mitoBEs展现出卓越的链特异性和显著降低的脱靶效应。得益于其双向碱基编辑能力,mitoBEs能够对大约87%的致病线粒体突变进行精确建模[7]。
2025年1月22日,昌平实验室及北京大学魏文胜课题组在Nature杂志在线发表了题为“Precise modelling of mitochondrial diseases using optimized mitoBEs”的研究论文。该研究报道了通过优化后的mitoBEs实现高效且精准地构建线粒体疾病小鼠模型的成果。利用优化版mitoBEs,研究团队成功建立了具有高突变频率的小鼠模型,这些模型表现出了与疾病相关的典型表型。此外,通过杂交实验,还获得了突变负荷达到100%以及仅含单碱基突变的精确小鼠模型。
为准确建立突变与疾病表型之间的直接联系,消除碱基编辑工具的脱靶效应尤为重要。在利用mitoBEs进行建模时,需要将RNA编码的mitoBEs注射到小鼠受精卵中。因此,该研究首先对RNA编码的mitoBEs系统的脱靶效应进行了全面评估。结果表明,RNA编码的mitoABE存在广泛的转录组脱靶效应,而mitoCBE则表现出一定程度的依赖于APOBEC1蛋白的线粒体基因组脱靶效应。为了提高mitoBEs的精准性,该研究重点优化了脱氨酶。针对mitoABE,通过突变筛选发现,TadA8e-V106W-V28F能够显著降低转录组脱靶至背景水平(图1)。针对mitoCBE,筛选了多种现有的胞嘧啶脱氨酶,并发现TadA衍生的胞嘧啶脱氨酶CBE6d在线粒体基因组上表现出的脱靶效应接近背景水平。基于这些优化成果,研究团队将改进后的mitoBEs命名为mitoBEs v2,包括mitoABE v2和mitoCBE v2(图1)。此外,该研究还系统性地评估了优化前后mitoBEs在核基因组上的脱靶效应,结果显示,无论是优化前还是优化后的mitoBEs,均未在核基因组上引发明显的脱靶效应,从而验证了其在基因编辑中的安全性和可靠性。
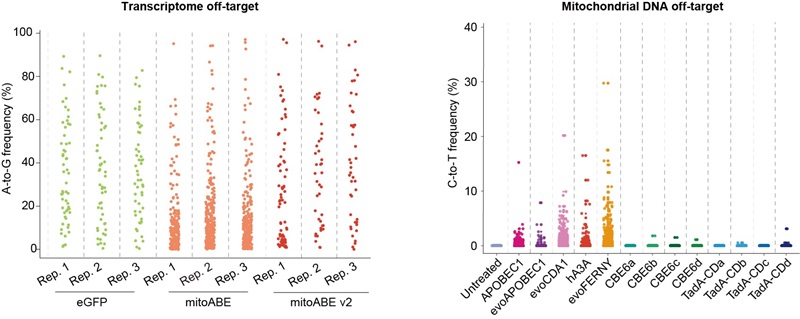
图1 优化mitoBEs的编辑精准性
通过将85个人类致病性线粒体DNA点突变与小鼠线粒体基因组进行同源性比对,研究确定了70个可编辑位点。进一步的细胞水平初步筛选成功实现了其中68个位点的编辑。比较发现,由环状RNA(circRNA)编码的mitoBEs v2相比于mRNA编码的工具,具有更高的编辑效率。因此,研究团队将circRNA编码的mitoBEs v2注射至小鼠胚胎并进行移植,结果显示mitoBEs v2在多种F0代小鼠模型中均实现了较高的编辑效率,其中mt-Nd5 A12784G F0小鼠模型的突变频率高达82%(图2)。此外,该研究系统性评估了F0代小鼠在线粒体基因组和核基因组中的脱靶效应,结果表明,在整个基因组范围内未检测到脱靶效应。这一发现表明,mitoBEs v2能够构建遗传背景干净的线粒体疾病小鼠模型。更重要的是,线粒体基因组的编辑结果在小鼠不同组织中表现出广泛且持久的存在(图2),并且能够通过母系遗传稳定传递。通过进一步杂交实验,研究成功获得了目标位点编辑效率达到100%以及仅含目标位点突变的mt-Nd5 A12784G小鼠模型。
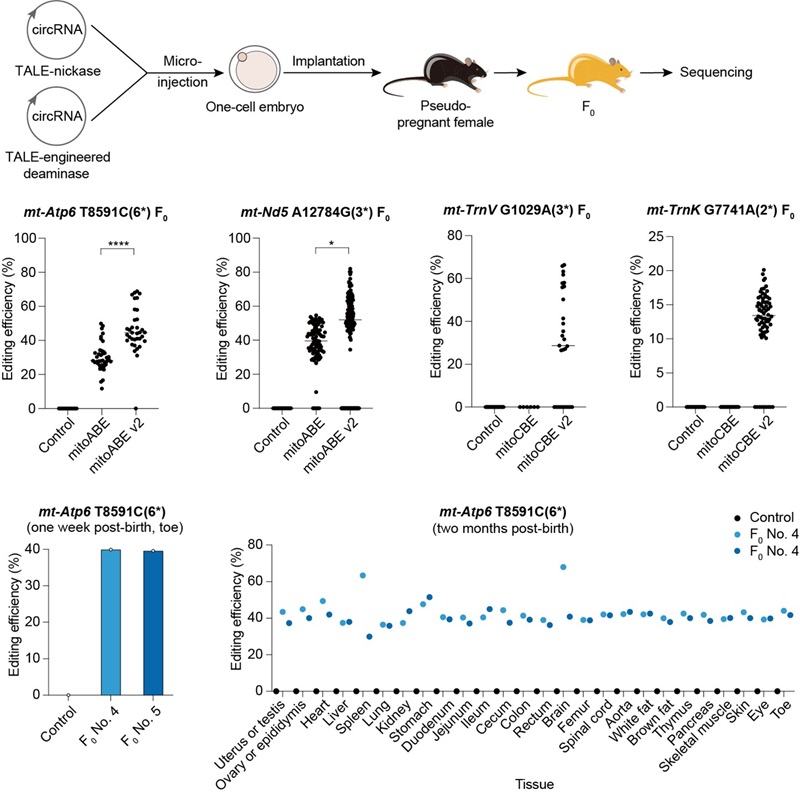
图2 mitoBEs v2高效构建线粒体疾病小鼠模型,编辑结果广泛且持久稳定于多组织
mt-Atp6 T8591C和mt-Nd5 A12784G分别对应人类线粒体致病突变m.T9191C和m.A13379G,并分别导致Leigh综合征和LHON。研究团队对突变率较高的F0代小鼠进行了疾病表型评估,结果显示,mt-Atp6 T8591C小鼠表现出显著的心脏功能障碍,与Leigh综合征的临床特征相符;mt-Nd5 A12784G小鼠则表现出类似LHON的视力障碍(图3)。此外,研究还通过调整TALE结合位点,成功构建了仅含目标位点编辑的单碱基突变mt-Nd5 A12784G小鼠模型。这些研究结果充分证明了mitoBE v2在创建线粒体疾病小鼠模型方面的高效性和精准性,为深入探索线粒体疾病的致病机制及开发新型治疗策略提供了重要工具。
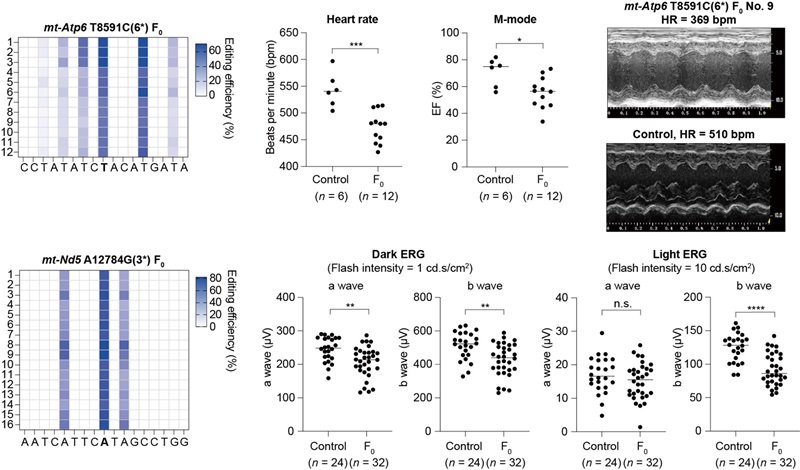
图3 小鼠模型呈现出相应的线粒体疾病表型
北京大学博士后伊宗裔为该论文的共同通讯作者,昌平实验室博士后张小雪为论文的第一作者,张雪、任纪武、李佳怡、魏晓旭和于莹博士也为该研究做出了重要贡献。本研究得到了昌平实验室、国家自然科学基金、北大-清华生命科学联合中心及中国博士后科学基金的资助。
Cell Reports|魏文胜团队利用碱基编辑器筛选绘制DNA损伤应答功能元件图谱
2024.12.13DNA损伤修复在维持基因组稳定性中发挥关键作用,其相关基因的突变可能危害基因组完整性,并增加癌症发生的风险。顺铂(Cisplatin)和电离辐射(IR)等DNA损伤药物被广泛用于肿瘤治疗,但临床治疗中不断发现病人对这些治疗产生耐药性突变。
2024年12月11日,北京大学魏文胜团队在Cell Reports杂志在线发表题为“Mapping functional elements of the DNA damage response through base editor screens”的研究论文。基于实验室此前开发的赖氨酸功能位点筛选策略和基因敲除策略1–3,研究团队分别利用腺嘌呤碱基编辑器和胞嘧啶碱基编辑器,对全基因组范围内的赖氨酸位点或基因进行靶向编辑,实现赖氨酸位点突变或敲除基因。随后在人类视网膜色素上皮细胞系(RPE1)中进行筛选,并结合两种DNA损伤药物处理,最终筛选出多个在DNA损伤修复过程中具有关键作用的氨基酸位点及基因(图1)。
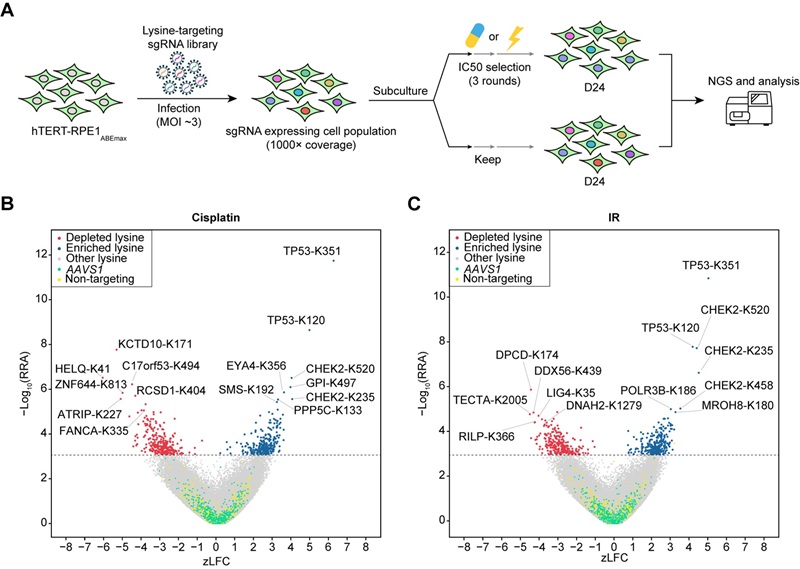
图1 筛选流程和结果
在Cisplatin筛选结果中,负向排名前十的基因大多为已知FA修复通路的关键因子(如FANCM、FANCA、FANCG等)。研究发现其中STK35基因从未被发现与DNA损伤应答相关。文献显示,其家族成员STK19和STK11分别参与TC-NER途径和UVB诱导损伤修复。后续实验证实,STK35是一个潜在的DNA修复因子,在DNA修复过程中发挥重要作用。
2019年,C17orf53被鉴定为DNA链间交联(ICL)修复的关键因子。在本研究中,筛选结果表明C17orf53的K494位点是其在DNA损伤修复中的关键氨基酸位点。K494的突变破坏了C17orf53与其上游因子RPA的相互作用,导致严重的ICL修复缺陷、G2/M细胞周期停滞以及对顺铂治疗的敏感性显著增强。
此外,研究还鉴定出多个与DNA损伤修复相关的重要翻译后修饰位点,例如p53的K120是调控其凋亡功能的重要乙酰化位点,该位点突变可导致细胞对DNA损伤药物的高度耐受。
总结来说,本研究通过多种碱基编辑筛选策略,系统性地绘制了影响DNA损伤应答的功能性赖氨酸位点及相关基因图谱,为DNA损伤应答基因功能研究提供了全新视角,也有助于加速癌症治疗中的耐药性变异研究(图2)。
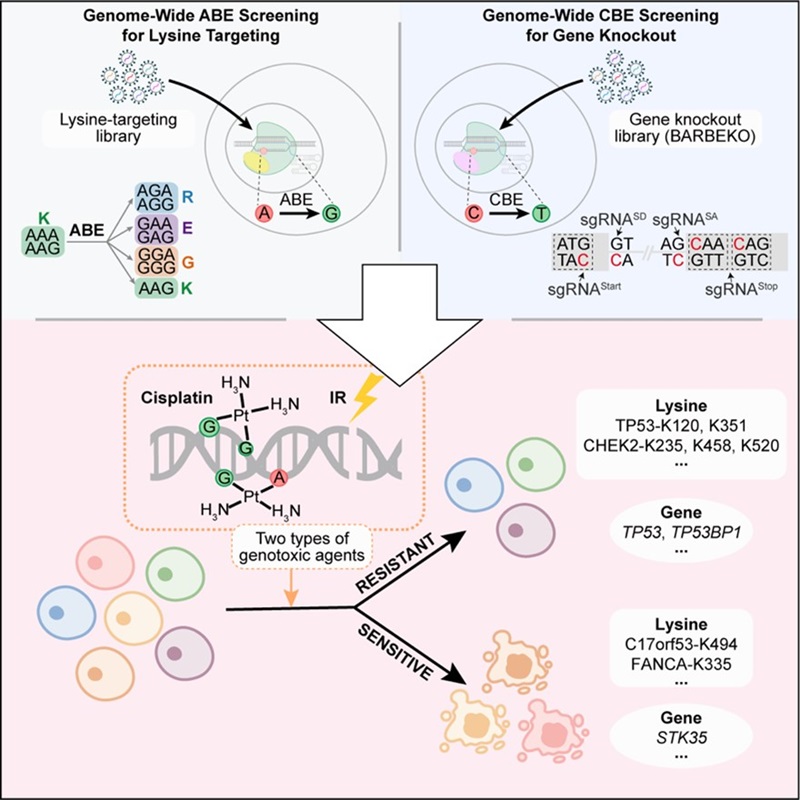
图2 总结图
本研究的共同第一作者为北京大学魏文胜课题组的潘倩博士(已毕业)与博士研究生张芷瑄。博士研究生熊杨芳、博士后宝颖、博士研究生陈天欣和许萍博士(已毕业)等人亦作出了重大贡献。研究获得了国家自然科学基金、北大-清华生命科学联合中心及昌平实验室的资助。
文章链接: https://doi.org/10.1016/j.celrep.2024.115047.
参考文献:
1. Xu, P., Liu, Z., Liu, Y., Ma, H., Xu, Y., Bao, Y., Zhu, S., Cao, Z., Wu, Z., Zhou, Z., et al. (2021). Genome-wide interrogation of gene functions through base editor screens empowered by barcoded sgRNAs. Nat Biotechnol 39, 1403–1413. https://doi.org/10.1038/s41587-021-00944-1.
2. Zhu, S., Cao, Z., Liu, Z., He, Y., Wang, Y., Yuan, P., Li, W., Tian, F., Bao, Y., and Wei, W. (2019). Guide RNAs with embedded barcodes boost CRISPR-pooled screens. Genome Biology 20. https://doi.org/10.1186/s13059-019-1628-0.
3. Bao, Y., Pan, Q., Xu, P., Liu, Z., Zhang, Z., Liu, Y., Xu, Y., Yu, Y., Zhou, Z., and Wei, W. (2023). Unbiased interrogation of functional lysine residues in human proteome. Molecular Cell 83, 4614-4632.e6. https://doi.org/10.1016/j.molcel.2023.10.033.
Nature Chemical Biology | 魏文胜团队实现蛋白质组中丝氨酸、苏氨酸和酪氨酸位点的功能解析
2024.09.23氨基酸是组成蛋白质的基本单位,其突变可以通过多种方式影响蛋白质功能,进而影响细胞生存和多种疾病的发生发展。此前研究已证明,使用碱基编辑器进行内源性氨基酸替换,并结合细胞筛选来大规模研究其功能的可行性1–3。磷酸化是细胞中常见且重要的翻译后修饰,绝大多数发生在丝氨酸、苏氨酸和酪氨酸位点上。目前已报道的大规模鉴定这三种氨基酸功能的研究主要依赖于生物信息学预测4或仅聚焦于已被检测到可发生磷酸化的位点5。考虑到蛋白组中依然存在大量潜在的磷酸化位点,且这三种氨基酸也介导了其它多种重要功能,实现在全蛋白组水平对这三种氨基酸位点的大规模功能鉴定具有重要意义。
2024年9月23日,北京大学魏文胜团队在Nature Chemical Biology在线发表题为“Functional profiling of serine, threonine, and tyrosine sites”的研究论文。研究基于实验室此前开发的赖氨酸功能位点筛选策略3,利用腺嘌呤碱基编辑器对全基因组范围内的丝氨酸、苏氨酸和酪氨酸位点进行靶向编辑,并在人类视网膜色素上皮细胞系(RPE1)中进行了细胞筛选,成功鉴定出3,467个功能性氨基酸位点。这些位点的突变展示了与基因敲除类似或不同的表型,尤其是大量功能性位点位于敲除不影响细胞生长的基因上,展示了从单个氨基酸层面绘制蛋白组功能图谱的重要性(图1)。

图1 蛋白质组功能性丝氨酸、苏氨酸和酪氨酸筛选示意图
接着,研究者对筛选出的功能性位点进行深入分析,特别关注磷酸化修饰。结果显示,677个筛选到的位点可能通过磷酸化修饰影响细胞生长,其中绝大多数为之前未知的功能性磷酸化位点(图2)。进一步研究表明,一些位点突变通过调节磷酸化水平导致信号通路异常,如MAPK信号通路中的MAPKAPK2 T338A突变以及MAP2K1 Y130H和S194P突变,影响了细胞的生长。

图2 筛选鉴定出大量功能性磷酸化位点
此外,研究者还分析了突变在蛋白质结构中的分布,发现丝氨酸到脯氨酸的替换广泛影响不同类型的蛋白质结构域功能,尤其是WD重复结构域中的两个高度保守的丝氨酸,暗示这些位点在维持蛋白质结构方面具有关键作用。
最后,研究发现,筛选出的促进细胞生长的突变与临床癌症患者的基因数据存在高度关联性(图3)。由于RPE1细胞系是永生化的正常细胞系,得到的309个可促进细胞生长的突变均可能为潜在的癌症驱动突变。据此猜想,研究者选择了MAP4K4-Y210C进行了体外克隆形成和小鼠体内成瘤实验,结果均证实了MAP4K4-Y210作为癌症驱动突变的潜力。

图3 与癌症临床数据相关的正向富集氨基酸位点
总体而言,该研究绘制了人类蛋白组中丝氨酸、苏氨酸和酪氨酸位点的功能图谱,揭示了这些位点与磷酸化修饰、蛋白质结构维持及癌症相关突变的密切联系,为生物学机制研究和临床应用提供了重要指导(图4)。

图4 研究总结示意图
本研究由北京大学/昌平实验室魏文胜团队完成,博士后李依舟、已出站的博士后徐涛及已毕业的马华峥博士为共同第一作者。研究获得了国家自然科学基金、昌平实验室和北大-清华生命科学联合中心的支持。
文章链接:https://www.nature.com/articles/s41589-024-01731-0
2. Hanna, R. E. Massively parallel assessment of human variants with base editor screens.
Nat Commun|魏文胜团队报道非脱氨酶依赖的嘧啶碱基编辑器TBE
2024.08.02DNA碱基编辑工具的进步为疾病治疗带来了巨大的希望,可修复由单核苷酸多态性(SNPs)【1】引起的单基因遗传疾病。目前,胞嘧啶碱基编辑器和腺嘌呤碱基编辑器主要依靠脱氨酶将胞嘧啶 (C) 或腺嘌呤 (A) 转化为尿嘧啶 (U) 或肌苷 (I),脱氨后的U和I会分别被识别为胸腺嘧啶 (T) 和鸟嘌呤 (G),从而促进C-to-T和A-to-G碱基变化【2-4】。在此基础上,进一步引入DNA糖基化酶切割中间产物U或I,产生无尿嘧啶/无嘧啶位点(AP位点),可以实现碱基的颠换,包括C-to-G转换的CGBE【5、6】和A-to-T/C转换的AYBE【7、8】。但以上碱基编辑工具均依赖脱氨酶,限制了它们编辑T和G的能力,并且由于脱氨酶的过表达会引起一定的脱靶效应。
2024年7月30日,北京大学魏文胜课题组在Nature Communications杂志在线发表题为 “Programmable DNA pyrimidine base editing via engineered uracil-DNA glycosylase”的研究论文,报道了一种名为TBE (Thymine base editor) 的不依赖脱氨酶的DNA碱基编辑工具。与现存的其他胸腺嘧啶编辑工具相比,TBE表现出更高的编辑效率、更小的细胞毒性和更低的脱靶现象。

图1: 不依赖于脱氨酶的碱基编辑器
研究人员关注到人源化尿嘧啶DNA-糖基化酶 (hUNG) 的两个突变体可以在体外实现对胞嘧啶和胸腺嘧啶的直接切割【10】。因此,通过将hUNG突变体与Cas9蛋白结合,sgRNA靶向编辑位点可以在体内实现对DNA序列中的C和T的直接编辑 (图1),其中在报告系统上编辑C的效率可达到30%,与现存的CGBE编辑效率类似,但针对T的hUNG突变体编辑效率较低。
为了进一步提高胸腺嘧啶的编辑效率,研究人员通过对UNG结构理性设计、同源蛋白检索、定向进化筛选的策略找到了来自耐辐射奇球菌 (Deinococcus radiodurans) 的尿嘧啶DNA-糖基化酶 (DrUNG) 突变体可以在多个人类基因组位点上实现高效的胸腺嘧啶编辑 (图2),编辑结果显示平均T-to-C, T-to-G及T-to-A的比例为52%, 30%和18%。通过优化连接氨基酸、引入具有合成倾向性的DNA聚合酶,还可以进一步提高编辑效率与编辑纯度。全面评估显示,TBE在基因组或转录组水平上都不会导致严重的脱靶编辑,这表明其具有高度的编辑特异性。

图2: TBEs在人类基因组多个内源位点实现高效的编辑
近期,多个团队也报道了基于人源化糖基化酶的胸腺嘧啶碱基编辑器【11-12】。通过比较32个内源位点编辑结果,研究人员发现与基于人源的胸腺嘧啶碱基编辑器相比,TBE展现出更高的编辑效率。此外,细胞毒性实验也表明TBE具有更小的细胞毒性。
最后,通过将DrUNG突变体的mRNA与nickase SpCas9融合蛋白和sgRNA共转染到原代成纤维细胞中的α-L-艾杜糖醛酸酶缺陷细胞中,可以观察到该酶活性的恢复,证明了TBEs在相关疾病治疗方面的潜在应用前景。
北京大学/昌平实验室魏文胜课题组博士后伊宗裔、博士后张小雪、博士研究生魏晓旭、本科生李佳怡(2024级研究生新生)为论文的共同第一作者。本研究获得了国家自然科学基金,昌平实验室,北大-清华生命科学中心及中国博士后科学基金资助。
文章链接:https://www.nature.com/articles/s41467-024-50012-w
1. Porto, E.M., Komor, A.C., Slaymaker, I.M. & Yeo, G.W. Base editing: advances and therapeutic opportunities. Nat Rev Drug Discov 19, 839-859 (2020).
2. Anzalone, A.V., Koblan, L.W. & Liu, D.R. Genome editing with CRISPR-Cas nucleases, base editors, transposases and prime editors. Nat Biotechnol 38, 824-844 (2020).
3. Gaudelli, N.M. et al. Programmable base editing of A*T to G*C in genomic DNA without DNA cleavage. Nature 551, 464-471 (2017).
4. Komor, A.C., Kim, Y.B., Packer, M.S., Zuris, J.A. & Liu, D.R. Programmable editing of a target base in genomic DNA without double-stranded DNA cleavage. Nature 533, 420-424 (2016).
5. Zhao, D. et al. Glycosylase base editors enable C-to-A and C-to-G base changes. Nat Biotechnol 39, 35-40 (2021).
6. Kurt, I.C. et al. CRISPR C-to-G base editors for inducing targeted DNA transversions in human cells. Nat Biotechnol 39, 41-46 (2021).
7. Tong, H. et al. Programmable A-to-Y base editing by fusing an adenine base editor with an N-methylpurine DNA glycosylase. Nat Biotechnol 41, 1080-1084 (2023)
8. Chen, L. et al. Adenine transversion editors enable precise, efficient A*T-to-C*G base editing in mammalian cells and embryos. Nat Biotechnol (2023)
9. Tong, H. et al. Programmable deaminase-free base editors for G-to-Y conversion by engineered glycosylase. Natl Sci Rev 10, nwad143 (2023).
10. Parikh, S.S., Mol, C.D., Slupphaug, G., Bharati, S., Krokan, H.E. & Tainer, J.A. Base excision repair initiation revealed by crystal structures and binding kinetics of human uracil-DNA glycosylase with DNA. EMBO J 17, 5214-5226 (1998).
11. Ye, L. et al. Glycosylase-based base editors for efficient T-to-G and C-to-G editing in mammalian cells. Nat Biotechnol (2024).
12. He, Y. et al. Protein language models-assisted optimization of a uracil-N-glycosylase variant enables programmable T-to-G and T-to-C base editing. Mol Cell (2024).
今夏记忆留江畔,青春足迹遍山城 | Wei Lab 2024年毕业旅行
2024.07.23又是一年盛夏时节,又是一年同时属于收获与告别的毕业季。而在旅行远足中放松自我同时拥抱下一阶段的新生活,已经成为了Wei Lab的传统。2024年,经实验室集体投票选择,位于祖国大西南的重庆市成为了本年度Wei Lab毕业旅行的目的地。
7月12日,大家陆续到达重庆市区。这一日的自由行给予了大家足够的时间沉浸式体验山城风情。大家或震撼于朝天门广场两江汇流的壮观,或醉心于洪崖洞流光溢彩的灯火夜景;或沉迷于重庆火锅的热辣滚烫,或流连于鹅岭贰厂的文化潮流;或在金色印象的舒适按摩中一扫疲惫,或在江畔酒吧的杯盏交错间品味人生。
7月13日一早,大家乘车前往地属武陵山脉的仙女山国家森林公园。在地处南国的重庆能看到宛如塞北的高山草原也是令人眼前一亮。大家奔跑在轻柔的绿草间,在风中吹出一串串七彩的气泡,那是飞舞着的彩色梦想,是跃动着的闪耀青春。下午,大家到达仙女山与武隆县城之间天生三桥景区,一览天生石桥的气势磅礴,山间的林森木秀与飞泉流瀑。晚上夜宿武隆县城,充满着烟火气息的烧烤与啤酒为脚步匆匆的一日行程作结。

7月14日清晨,大家乘车到达位于酉阳的龚滩古镇,走在石板街上,穿过吊脚楼间,品味浓浓的古镇风情。午饭后,实验室全体乘船泛舟乌江,沿乌江画廊至彭水县城。江水碧绿,江风阵阵,我们的欢声笑语也随轻舟一起飘过万重山。下午乘竹筏在阿依河上对唱,歌声在山谷里回荡。晚间在民宿外围着篝火跳舞,燃烧的火苗照亮了每一张快乐的面庞。

7月15日是出行的最后一日,大家起大早前往隔摩围山与彭水老城区相邻的蚩尤九黎城,深度体验了神秘的苗家文化。在品尝了苗家长桌宴的特色后,大家乘车返回重庆市区,在解放碑商业步行街狂欢了最后一个下午,于暮色中挥别山城,乘坐飞机返回了北京。
人世间也有太多如山城和古镇里一样崎岖弯折的路,但无论山高水深,它们都通往终点的灯火灿烂。我们在路上相逢,并一起写下属于青春的美丽故事。毕业不是终点,未来永远可期。
今年也是Wei Lab成立十七周年,大家在蚩尤九黎城前也拉出了庆祝的横幅。无论是过去还是今朝的我们永远是基因编辑领域的风流人物,也祝福Wei Lab的前沿“文”章常出新,无限“胜”景在险峰!

供稿人:牛煦然
2024毕业快乐
2024.07.02祝贺潘倩、田峰和拉毛切忠同学获得博士学位!祝贺陈鑫鹏、李帛翰、李佳怡、龙语纯、苏洪波和王璐同学获得学士学位!同时,祝贺潘倩、拉毛切忠、李帛翰和苏洪波同学获得“北京大学优秀毕业生”称号。
毕业是一个新的开始,愿你们在新的起点上勇敢地迈出第一步,开启属于自己的精彩人生!



祝贺牛煦然同学获得2024-2025学年校长奖学金
2024.06.132024年6月,根据《北京大学博士研究生校长奖学金管理办法》及相关规定要求,经过生命科学学院评审委员会组织的答辩和评审工作,Wei Lab 2020级研究生牛煦然获得“北京大学2024-2025学年博士研究生校长奖学金”。
校长奖学金是北京大学设立的荣誉性最高、资助额度最大、影响范围最广的研究生奖学金,旨在优化高层次人才培养环境,激励博士生从事高水平科学研究,促进学校的学科发展和研究生培养质量的全面提高。
祝贺牛煦然同学获得2024年瑞沃德奖学金
2024.04.232024年4月,经自主申请和北京大学生命科学学院组织答辩,Wei Lab 2020级研究生牛煦然获得“2024年北京大学生命科学学院瑞沃德奖学金”。
本年度的瑞沃德奖学金答辩是该奖项设立以来的首届评选。学院共收到40余份自主报名材料,从中遴选出9位参评者进行统一答辩,并最终评选出5位获奖人。2024年4月23日,在吕志和楼进行了奖学金颁奖仪式。

(左起第五位为牛煦然同学)
附:
瑞沃德奖学金简介
深圳市瑞沃德生命科技股份有限公司(简称:瑞沃德)成立于2002年,是一家专业服务于生命科学、动物健康以及临床医疗领域的国家高新技术企业。为支持北京大学生命科学学院教育事业的发展,深圳市瑞沃德生命科技有限公司于2023年捐资设立北京大学生命科学学院“瑞沃德奖学金”项目,每年评选5名获奖人,旨在激励优秀研究生积极进取、刻苦钻研、不断创新,成为未来科研领域的佼佼者,以助力国内生物学科研人才培养,推动生物学科建设和发展。
Cellular & Molecular Immunology | 揭示肿瘤逃逸非HLA-I类分子依赖多效型T细胞杀伤的新机制
2024.02.21在生物体演化和优化的过程中,多细胞生物逐渐进化出了"枪杆子",即效应杀伤细胞群,以极其有效的方式识别和杀伤有病变的细胞。肿瘤的发生和发展需要逃避效应细胞的免疫监管。那么,肿瘤细胞是如何逃逸效应细胞攻击的呢?
2024年2月20日,Cellular & Molecular Immunology发表了来自北京大学魏文胜团队的最新科研成果,题为《Unsynchronized butyrophilin molecules dictate cancer cell evasion of Vγ9Vδ2 T-cell killing》,揭示了一系列肿瘤逃逸Vγ9Vδ2 T细胞的分子机制。

Vγ9Vδ2 T细胞是人外周血中最主要的一类γδ T细胞,具备强大而广泛的肿瘤杀伤能力,是免疫疗法中极具潜力的底盘细胞。一方面,Vγ9Vδ2 T细胞通过其TCR识别肿瘤细胞表面的人嗜乳糜蛋白(BTN)分子,清除磷抗原水平失衡的细胞,实现对肿瘤的清除,而无需依赖HLA分子。另一方面,Vγ9Vδ2 T细胞具有天然免疫细胞的功能特征,即通过天然杀伤细胞(NK)活化受体来杀伤靶细胞。
研究利用全基因组CRISPR功能筛选,确定了调控Vγ9Vδ2 T细胞杀伤肿瘤细胞的上下游因子。通过在代表实体瘤和液体瘤的细胞系中进行实验,发现敲除包括BTN2A1、BTN3A1、BTN3A2、BTN3A3、QPCTL在内的两百多个基因会导致肿瘤对Vγ9Vδ2 T细胞介导的杀伤逃逸(如下图所示)。研究还发现,在一千余种肿瘤细胞系中,BTN分子呈现共表达的趋势,并受到IFN-γ通路和RFX家族的调控。进一步的研究表明,在B2M(HLA分子的组成部分)低表达的肿瘤中,BTN分子可以独立预测临床结局。这意味着Vγ9Vδ2 T细胞和αβT细胞在抗肿瘤效应上发挥独立且协同的作用。
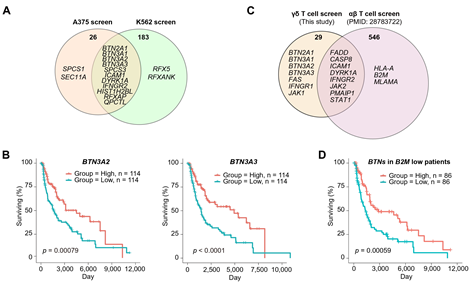
图1. Vγ9Vδ2 T细胞杀伤筛选结果
研究首次鉴定了BTN分子的一种蛋白质翻译后修饰,即N端谷氨酰胺(Gln)的焦谷氨酸化(pyroglutamate)修饰。研究发现,QPCTL是BTN分子发生焦谷氨酸化修饰的关键酶。当QPCTL基因被敲除时,BTN分子的N端谷氨酰胺无法发生焦谷氨酸化修饰,从而影响Vγ9Vδ2 T细胞对肿瘤细胞的识别和杀伤(如下图所示)。
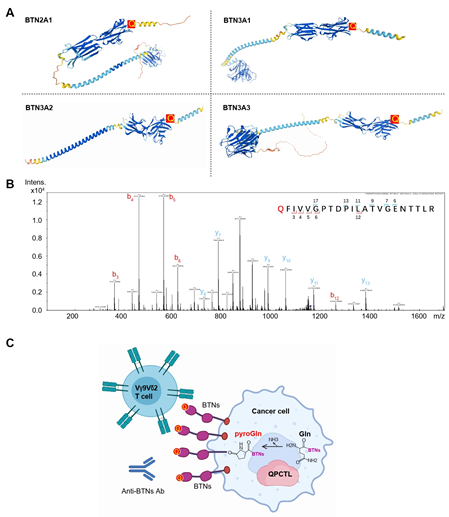
图2. BTN分子N端谷氨酰胺的焦谷氨酸化修饰
该研究揭示了BTN分子在序列进化水平、转录水平、表达水平和蛋白质翻译后修饰水平上的一致性及功能鲁棒性。并表明BTN3A2和BTN3A3分子也参与T细胞的活化。最新研究表明BTN3A1分子与TCR之间存在相互作用(bioRxiv 2023.08.30.555639)。BTN3A1分子和TCR分子的互作位点与BTN2A1和BTN3A1分子之间的互作位点相一致。在正常细胞中,BTN分子之间形成了"活化表位遮蔽"状态。当BTN分子之间的相互作用受到干扰时,可以通过两种方式来实现:一是通过异位抗体的结合(由外而内),二是通过磷抗原的作用(由内而外),从而激活T细胞。该研究提示BTN3A2和BTN3A3参与了表位遮蔽和TCR活化的生物学过程。
随着Vγ9Vδ2 T细胞在肿瘤免疫领域应用的不断拓展,诱导和增强γδ T细胞对肿瘤细胞和感染细胞的识别和杀伤变得至关重要。其中,临床上使用唑来膦酸来激活T细胞已成为一种重要策略。该研究系统鉴定了介导唑来膦酸入胞的效应分子(如下图所示),有助于深入了解其作用机制并开发相关的治疗药物。此外,鉴定与Vγ9Vδ2 T细胞效应功能相关的增效基因,有利于推动细胞基因疗法的研究,提高抗瘤效能。
图3. 唑来膦酸入胞效应分子及BTN分子活化关键步骤
北京大学魏文胜教授为论文主要通讯作者,北京协和医院黄超兰教授为共同通讯作者。北京大学生物医学前沿创新中心武泽光博士、拉毛切忠博士(CLS毕业生)和顾美超博士为共同第一作者。本研究还得到了课题组博士生靳宣宣、刘莹博士, 墨尔本大学A. Uldrich博士以及博雅辑因袁鹏飞博士的帮助和宝贵建议。该研究获得了国家自然科学基金、北大-清华生命科学联合中心、昌平实验室等的支持。
祝贺拉毛切忠同学获得博士学位
2023.12.15祝贺拉毛切忠同学顺利通过学位论文答辩,于2024年春季获得博士学位!同时,祝贺拉毛切忠同学获得“北京大学优秀毕业生”的荣誉!期待你在未来的道路上继续砥砺前行,成为最好的自己!


Mol Cell|魏文胜团队实现人类蛋白质组中赖氨酸位点的功能解码
2023.11.23氨基酸作为构成蛋白质的基本单位,对于蛋白质的结构和功能至关重要,而其变化也与多种疾病的发生发展密切相关。尽管目前利用碱基编辑等技术1,2可以在基因组中实现碱基替换进而改变密码子,产生内源氨基酸的突变,但在全蛋白质组范围内对特定氨基酸残基进行系统性的功能分析仍然面临挑战。
2023年11月22日,北京大学魏文胜团队在Molecular Cell杂志在线发表了题为“Unbiased interrogation of functional lysine residues in human proteome”的研究论文。该研究采用腺嘌呤碱基编辑工具,建立了一种在全蛋白质组范围内筛选功能性氨基酸位点的策略,并通过细胞适应性筛选,获得了蛋白质组功能性赖氨酸位点的图谱。
在人类蛋白质组众多的氨基酸残基中,研究者首先关注了赖氨酸。赖氨酸残基携带正电荷,在蛋白质的结构、与其他分子的相互作用等方面发挥着重要作用,同时也是多种蛋白质翻译后修饰(如泛素化、乙酰化、甲基化)的重要受体氨基酸残基。赖氨酸的密码子为5’-AAA或5’-AAG,利用腺嘌呤编辑器(Adenine base editors, ABEs)对其密码子进行编辑,可以实现赖氨酸的定向突变(图1)。
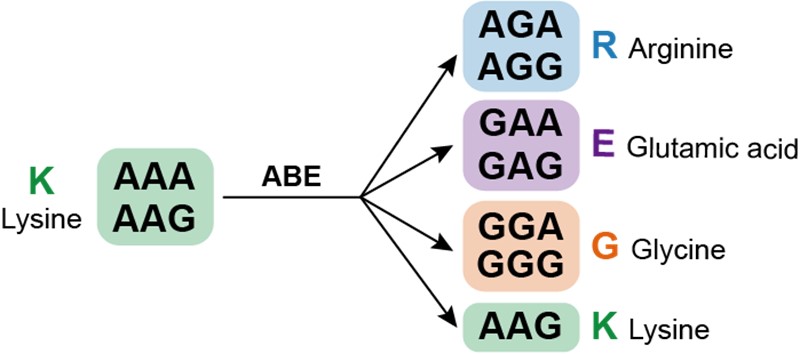
图1 赖氨酸定向突变示意图
该研究利用ABEmax系统在人视网膜色素上皮细胞系RPE1中构建了靶向赖氨酸位点的sgRNA文库3,包含约30万条sgRNA,覆盖了85%的编码基因、85%的蛋白质以及35%的赖氨酸密码子。为提高筛选质量并大幅缩减建库所需细胞数量,研究者结合团队前期建立的iBAR策略进行了细胞适应性筛选4,最终获得了1572个促进或抑制细胞存活的赖氨酸突变位点。
基于课题组前期在RPE1细胞系中进行的基因敲除筛选结果5,研究者将赖氨酸位点的得分映射到其所在基因上,形成了基因(蛋白)-赖氨酸位点的功能性图谱(图2)。值得注意的是,大量赖氨酸位点的突变导致细胞表现出与相应基因敲除不同的细胞适应性表型。在这些突变位点中,有805个位点突变后会抑制细胞存活,然而对应的基因敲除却未对细胞存活产生影响或促进细胞存活。通过对国际癌症基因组联盟数据库(ICGC)的数据挖掘,研究者鉴定了若干具有临床意义的赖氨酸突变位点,包括已有报道的TP53-K120、BRAF-K601、PTEN-K13等位点,然而大多数赖氨酸位点的功能仍不为人知。
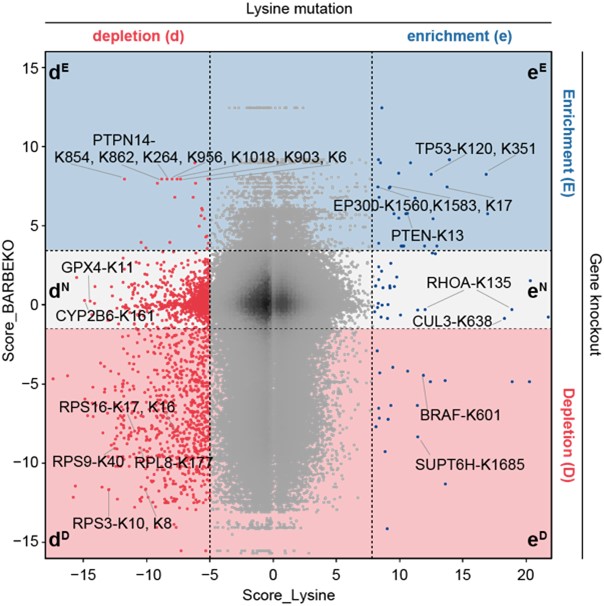
图2 两种筛选结果对比图
在正向富集的赖氨酸位点中,研究者发现了一个以CUL3为核心的调控网络。CUL3是泛素化复合物(cullin-RING ligases,CRLs)中的重要成员。该研究筛选到位于其骨架蛋白、接头蛋白、激活蛋白、底物蛋白多个赖氨酸位点(图3)。通过亲和纯化-质谱等方法,研究者发现CUL3-K638E能够显著削弱CUL3和去nedd化复合物(COP9 signalosome,CSN)的结合,导致其持续处于nedd化状态而最终降低了稳定性。
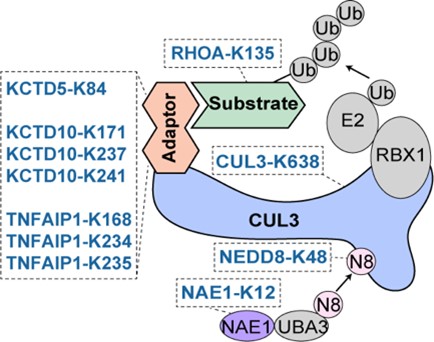
图3 CUL3 CRLs复合物
最终,研究者将关注点聚焦在CUL3的接头蛋白KCTD10的K171位点上,该位点在一名乳腺癌患者体内检测到了突变(K171E)。通过一系列实验,研究者证实KCTD10-K171能够发生乙酰化修饰,并通过调控细胞周期蛋白TPX2和INCENP的蛋白稳态来控制下游信号通路,确保细胞周期正常进行。KCTD10-K171的突变可能导致细胞周期蛋白无法正常降解,从而引起细胞过度增殖。该位点可能成为潜在的癌症标志突变,有助于癌症的诊断和预后,并为新药研发提供了指导。
总的来说,本研究利用碱基编辑技术成功建立了高通量的氨基酸精度功能性筛选方法,为系统性研究蛋白质功能和调控机制提供了新的有效手段。同时,所得到的氨基酸精度的蛋白质功能性大数据为更深入地理解蛋白质调控机制,尤其是翻译后修饰机制,提供了有力的依据(图4)。

图4 总结图
北京大学魏文胜课题组博士后(CPL)宝颖、博士研究生潘倩和已毕业的许萍博士和刘志恒博士为论文的共同第一作者,魏文胜教授和副研究员周卓(现为中国医学科学院系统医学研究院/苏州系统医学研究所研究员)为论文的共同通讯作者。该研究获得了国家重点研发计划、国家自然科学基金、北京市科委生物医学前沿创新推进项目、中国医学科学院医学与健康科技创新工程、北大-清华生命科学联合中心、昌平实验室、重大疾病共性机制研究全国重点实验室等的支持。
文章链接: https://doi.org/10.1016/j.molcel.2023.10.033.
参考文献:
1. Komor, A.C., Kim, Y.B., Packer, M.S., Zuris, J.A., and Liu, D.R. (2016). Programmable editing of a target base in genomic DNA without double-stranded DNA cleavage. Nature 533, 420–424. 10.1038/nature17946.
2. Gaudelli, N.M., Komor, A.C., Rees, H.A., Packer, M.S., Badran, A.H., Bryson, D.I., and Liu, D.R. (2017). Programmable base editing of A•T to G•C in genomic DNA without DNA cleavage. Nature 551, 464–471. 10.1038/nature24644.
3. Koblan, L.W., Doman, J.L., Wilson, C., Levy, J.M., Tay, T., Newby, G.A., Maianti, J.P., Raguram, A., and Liu, D.R. (2018). Improving cytidine and adenine base editors by expression optimization and ancestral reconstruction. Nat. Biotechnol. 36, 843–846. 10.1038/nbt.4172.
4. Zhu, S., Cao, Z., Liu, Z., He, Y., Wang, Y., Yuan, P., Li, W., Tian, F., Bao, Y., and Wei, W. (2019). Guide RNAs with embedded barcodes boost CRISPR-pooled screens. Genome Biol. 20. 10.1186/s13059-019-1628-0.
5. Xu, P., Liu, Z., Liu, Y., Ma, H., Xu, Y., Bao, Y., Zhu, S., Cao, Z., Wu, Z., Zhou, Z., et al. (2021). Genome-wide interrogation of gene functions through base editor screens empowered by barcoded sgRNAs. Nat. Biotechnol. 10.1038/s41587-021-00944-1.
Genome Biology | LEAPER 2.0在非人灵长类动物和人源化小鼠中实现了高效精准的长时RNA编辑
2023.10.25碱基编辑作为一项全新的疾病治疗策略,越来越受到广泛关注。ADAR作为一种细胞内源的RNA脱氨酶已经被广泛应用于RNA碱基编辑。基于ADAR的RNA编辑利用了细胞翻译机制的一个独特性质,即由于结构相似性,肌苷会被识别为鸟嘌呤。这使得ADAR编辑器能够引入特定位点的、由RNA引导的腺嘌呤到肌苷(A-to-I)的改变,从而打开了广泛的治疗潜力,包括修正致病突变、调节基因表达或改变蛋白质相互作用等[1]。2019年魏文胜课题组开发了LEAPER技术(Leveraging Endogenous ADAR for Programmable Editing of RNA)[2],并于2022年升级为LEAPER 2.0[3],通过工程化的环状RNA招募细胞内源的ADAR蛋白,在靶向位点实现了高效且精准的编辑。由于LEAPER是一种依赖于内源ADAR活性的编辑系统,而不同组织和物种中内源 ADAR 的表达存在差异,并且递送方法尚未得到全面验证,因此该技术仍需在多个物种,特别是非人灵长类中进行测试。在这项研究中,研究人员进一步利用AAV将工程化的环状ADAR招募RNA(circ-arRNA)递送到非人灵长类动物及人源化小鼠体内,以实现长期、高效和精准的RNA碱基编辑。
首先,研究人员在非人灵长类动物的体外培养细胞内进行了实验,通过优化获得了高效且精准的工程化circ-arRNA,其靶向位点的编辑效率可高达47%。随后,他们将该工程化circ-arRNA包装到AAV病毒中,成功实现了在非人灵长类动物体内的递送。然而,临床上使用的AAV剂量一旦超过1 × 1014 vg/kg,会带来严重毒性[4],因此该研究将AAV剂量控制在临床可接受的范围内,分别为3×1012,1×1013和3×1013 vg/kg的剂量 (图1A)。将这三种剂量的肝靶向AAV8注射到非人灵长类动物体内,并进行长期观察和检测,结果显示在第二周,肝脏细胞编辑效率可达50%以上(图1B)。通过计算感染效率(图1C)发现受AAV感染的肝脏细胞编辑效率可高达80%,这表明LEAPER 2.0可以在体内快速且高效地发挥作用。此外,研究人员还发现,编辑效率呈剂量依赖性,随着AAV8剂量的升高,非人灵长类动物体内工程化circ-arRNA的表达量增加,从而提高了编辑效率,这表明LEAPER 2.0具有良好的药物剂量依赖性 (图1D)。编辑效率在非人灵长类动物体内可以维持13周并保持稳定,说明通过AAV递送的LEAPER 2.0具有长期编辑的特点(图1E)。此外,研究者未观察到LEAPER 对非人灵长类动物具有毒性,表明该编辑技术是安全且特异的。
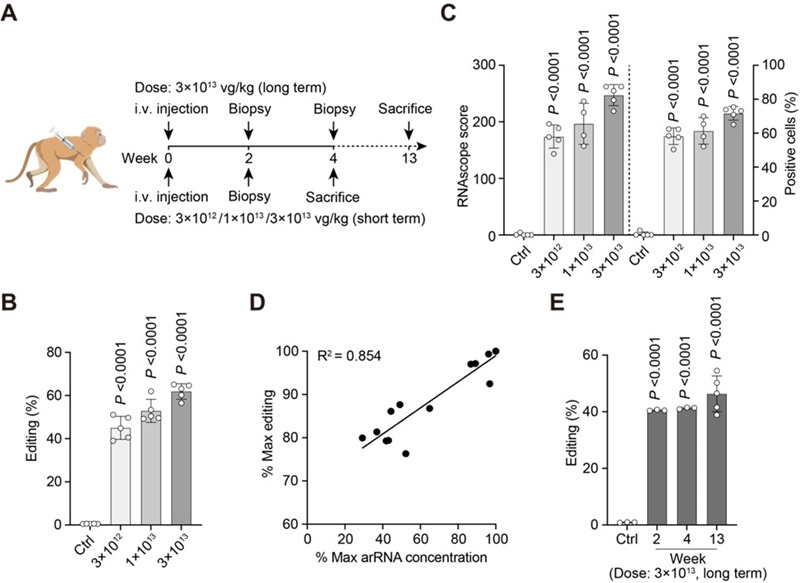
图1. AAV递送的LEAPER 2.0在非人灵长类动物体内实现高效、长时程编辑
进一步地,研究人员尝试使用AAV递送的LEAPER 2.0来治疗人源化转录本中的提前终止密码子。提前终止密码子导致了11%的人类遗传病[5],因此针对此类密码子的编辑具有巨大的应用前景。为此研究者构建了人源化的Hurler综合征小鼠,该小鼠表达人源化的IDUA转录本,其中存在一个提前终止密码子,导致该小鼠可以模拟人类溶酶体贮积症,即粘多糖贮积症 I 型 (MPS-I)(图2A)。 MPS-I的特征是α-L-艾杜糖苷酶的酶活性缺陷,导致糖胺聚糖 (GAG)的积累。通过筛选和理性设计,研究者成功地提高了ADAR编辑的精确性,并获得了高效的工程化circ-arRNA。使用AAV-PHP.eB递送circ-arRNA到小鼠体内,不仅在肝脏等器官,而且在神经系统恢复了α-L-艾杜糖苷酶的酶活性(图2B,C),降低了GAG的积累,显著改善了小鼠的表型。由于目前MPS-I的治疗策略多为酶替代疗法,递送的IDUA蛋白不能跨越血脑屏障,因此无法治疗中枢神经系统中的GAG积累[6],而LEAPER 2.0介导的RNA编辑能够在多个器官中恢复细胞内源IDUA转录本的功能,从而实现了更优的治疗效果。
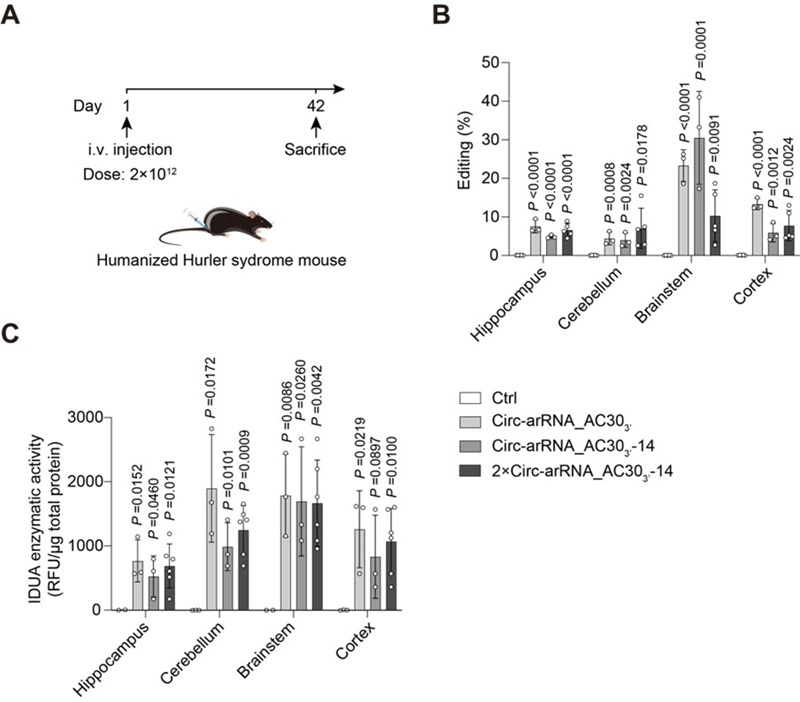
图2. AAV递送的LEAPER 2.0成功治疗人源化Hurler综合征小鼠
结合研究者在非人灵长类动物和人源化小鼠中的研究结果,LEAPER 2.0 为遗传性疾病治疗以及其他严重疾病的潜在临床应用提供了巨大的希望。这些发现极大地加强了通过AAV递送的工程化环状ADAR招募RNA用于治疗和探索性转化研究的前景。这项研究由博雅辑因生物科技有限公司和北京大学魏文胜课题组合作完成。博雅辑因科学创始人,北京大学魏文胜教授和博雅辑因袁鹏飞博士为该研究的共同通讯作者。魏文胜课题组伊宗裔博士、博雅辑因赵艳霞博士、易泽轩博士和张永建博士为论文的共同第一作者,博雅辑因汤刚彬博士等人也对这项研究作出了重要贡献。该研究项目得到了国家重点研发计划、国家自然科学基金重点项目、北京市科委生物医学前沿创新推进项目、北大-清华生命科学联合中心、昌平实验室和中国博士后科学基金的支持。
原文链接:https://genomebiology.biomedcentral.com/articles/10.1186/s13059-023-03086-6
参考文献:
1. Sheridan C: Shoot the messenger: RNA editing is here. Nat Biotechnol 2023, 41(3):306-308.
2. Qu L, Yi Z, Zhu S, Wang C, Cao Z, Zhou Z, Yuan P, Yu Y, Tian F, Liu Z et al: Programmable RNA editing by recruiting endogenous ADAR using engineered RNAs. Nat Biotechnol 2019, 37(9):1059-1069.
3. Yi Z, Qu L, Tang H, Liu Z, Liu Y, Tian F, Wang C, Zhang X, Feng Z, Yu Y et al: Engineered circular ADAR-recruiting RNAs increase the efficiency and fidelity of RNA editing in vitro and in vivo. Nat Biotechnol 2022, 40(6):946-955.
4. Agarwal S: High-dose AAV gene therapy deaths. Nat Biotechnol 2020, 38(8):910.
5. Mort M, Ivanov D, Cooper DN, Chuzhanova NA: A meta-analysis of nonsense mutations causing human genetic disease. Hum Mutat 2008, 29(8):1037-1047.
6. Parini R, Deodato F: Intravenous Enzyme Replacement Therapy in Mucopolysaccharidoses: Clinical Effectiveness and Limitations. Int J Mol Sci 2020, 21(8).
撰稿人:伊宗裔
日期:2023-10-23
图们江畔映笑语,长白山上留欢声 | Wei Lab 2023年毕业旅行
2023.07.247月14日,经过8个小时的高铁旅程,课题组所有成员到达了延边州的首府延吉市,开启了实验室2023年毕业旅行。7月15日,实验室全员乘车往返延吉和图们两市,在图们江畔的中朝边境口岸感受壮丽的国门风采,在延边博物馆了解延边州的历史文化,在朝鲜族民俗园领略朝鲜族同胞的风土人情,在延边大学网红弹幕墙下打卡延吉市的现代符号……伴着傍晚的清风,大家又乘车赶赴安图县,夜宿长白山下。

7月16日清晨,大家集体前往本次旅行最重要的一站:长白山。这一天的安图县晴雨间歇而至,而被长白山十六峰环抱簇拥的天池也在这一天悄然掩藏于浓雾之中,为本次旅行带来了一丝意犹未尽的体验感。好在半山处的长白瀑布、聚龙温泉、绿渊潭和小天池等景点也成为了大家快乐的休息打卡处。傍晚时分,大家从长白山乘车返回安图时,天空也最终放晴。从车上回望绵绵长白,又增添了几分朦胧的美。

人生也一直是晴雨间至,但是在彩虹的终点等待我们每个人的,也一定会是灿烂的晴天。人生这一路上也有很多的美可以去期待,有很多的快乐值得我们去体验,有很多的故事等待我们去一起参与和讲述。
长风破浪会有时,白驹过隙仍少年,山高海阔任君行。祝福Wei Lab 2023年的毕业生以及课题组的每一位成员都能在未来的人生路上乘风破浪前进,千帆过尽,归来仍是少年!
供稿人:牛煦然




